+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the human GBP1 dimer bound to GDP-AlF3 | |||||||||
 マップデータ マップデータ | Primary map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Cell-autonomous immunity / intracellular pathogens /  GTPase (GTPアーゼ) / GTPase (GTPアーゼ) /  IMMUNE SYSTEM (免疫系) IMMUNE SYSTEM (免疫系) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報GDP phosphatase activity / non-canonical inflammasome complex assembly / protein localization to vacuole / negative regulation of substrate adhesion-dependent cell spreading / symbiont cell surface /  cytolysis in another organism / positive regulation of pyroptotic inflammatory response / vesicle membrane / negative regulation of protein localization to plasma membrane / negative regulation of interleukin-2 production ...GDP phosphatase activity / non-canonical inflammasome complex assembly / protein localization to vacuole / negative regulation of substrate adhesion-dependent cell spreading / symbiont cell surface / cytolysis in another organism / positive regulation of pyroptotic inflammatory response / vesicle membrane / negative regulation of protein localization to plasma membrane / negative regulation of interleukin-2 production ...GDP phosphatase activity / non-canonical inflammasome complex assembly / protein localization to vacuole / negative regulation of substrate adhesion-dependent cell spreading / symbiont cell surface /  cytolysis in another organism / positive regulation of pyroptotic inflammatory response / vesicle membrane / negative regulation of protein localization to plasma membrane / negative regulation of interleukin-2 production / negative regulation of T cell receptor signaling pathway / cytolysis in another organism / positive regulation of pyroptotic inflammatory response / vesicle membrane / negative regulation of protein localization to plasma membrane / negative regulation of interleukin-2 production / negative regulation of T cell receptor signaling pathway /  spectrin binding / spectrin binding /  cytokine binding / defense response to protozoan / cytokine binding / defense response to protozoan /  加水分解酵素; 酸無水物に作用; リン含有酸無水物に作用 / cellular response to interleukin-1 / regulation of protein localization to plasma membrane / regulation of calcium-mediated signaling / G protein activity / 加水分解酵素; 酸無水物に作用; リン含有酸無水物に作用 / cellular response to interleukin-1 / regulation of protein localization to plasma membrane / regulation of calcium-mediated signaling / G protein activity /  lipopolysaccharide binding / lipopolysaccharide binding /  加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / 加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 /  Hsp90 protein binding / cytoplasmic vesicle membrane / negative regulation of ERK1 and ERK2 cascade / cellular response to type II interferon / GDP binding / Interferon gamma signaling / Hsp90 protein binding / cytoplasmic vesicle membrane / negative regulation of ERK1 and ERK2 cascade / cellular response to type II interferon / GDP binding / Interferon gamma signaling /  マイクロフィラメント / cellular response to tumor necrosis factor / マイクロフィラメント / cellular response to tumor necrosis factor /  actin binding / cytoplasmic vesicle / defense response to virus / defense response to bacterium / actin binding / cytoplasmic vesicle / defense response to virus / defense response to bacterium /  ゴルジ体 / ゴルジ体 /  自然免疫系 / 自然免疫系 /  GTPase activity / GTP binding / GTPase activity / GTP binding /  ゴルジ体 / ゴルジ体 /  enzyme binding / protein homodimerization activity / extracellular region / identical protein binding / enzyme binding / protein homodimerization activity / extracellular region / identical protein binding /  細胞膜 / 細胞膜 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
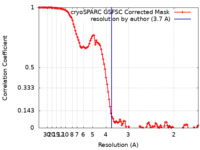
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.7 Å クライオ電子顕微鏡法 / 解像度: 3.7 Å | |||||||||
 データ登録者 データ登録者 | Kuhm TI / Jakobi AJ | |||||||||
| 資金援助 | European Union,  オランダ, 2件 オランダ, 2件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Cryo-EM structure of the human GBP1 dimer bound to GDP-AlF3 著者: Kuhm TI / de Agrela Pinto C / Taisne C / Huber S / Giannopolou N / Pardon E / Steyaert J / Jakobi AJ | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16794.map.gz emd_16794.map.gz | 127.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16794-v30.xml emd-16794-v30.xml emd-16794.xml emd-16794.xml | 23.6 KB 23.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16794_fsc.xml emd_16794_fsc.xml | 10.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16794.png emd_16794.png | 100.9 KB | ||
| マスクデータ |  emd_16794_msk_1.map emd_16794_msk_1.map | 134.6 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-16794.cif.gz emd-16794.cif.gz | 7.1 KB | ||
| その他 |  emd_16794_additional_1.map.gz emd_16794_additional_1.map.gz emd_16794_half_map_1.map.gz emd_16794_half_map_1.map.gz emd_16794_half_map_2.map.gz emd_16794_half_map_2.map.gz | 1.6 MB 124.9 MB 124.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16794 http://ftp.pdbj.org/pub/emdb/structures/EMD-16794 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16794 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16794 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8cqbMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16794.map.gz / 形式: CCP4 / 大きさ: 134.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16794.map.gz / 形式: CCP4 / 大きさ: 134.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Primary map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.834 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_16794_msk_1.map emd_16794_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
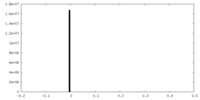
| 投影像・断面図 |
| ||||||||||||
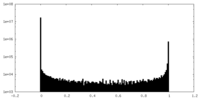
| 密度ヒストグラム |
-追加マップ: Locally sharpened map (LocScale)
| ファイル | emd_16794_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Locally sharpened map (LocScale) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
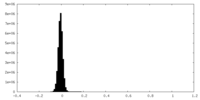
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_16794_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_16794_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Homodimeric complex of GBP1 stabilised by GDP-AlF3
| 全体 | 名称: Homodimeric complex of GBP1 stabilised by GDP-AlF3 |
|---|---|
| 要素 |
|
-超分子 #1: Homodimeric complex of GBP1 stabilised by GDP-AlF3
| 超分子 | 名称: Homodimeric complex of GBP1 stabilised by GDP-AlF3 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 134 KDa |
-分子 #1: Guanylate-binding protein 1
| 分子 | 名称: Guanylate-binding protein 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO EC番号:  加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 68.021617 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MASEIHMTGP MCLIENTNGR LMANPEALKI LSAITQPMVV VAIVGLYRTG KSYLMNKLAG KKKGFSLGST VQSHTKGIWM WCVPHPKKP GHILVLLDTE GLGDVEKGDN QNDSWIFALA VLLSSTFVYN SIGTINQQAM DQLYYVTELT HRIRSKSSPD E NENEVEDS ...文字列: MASEIHMTGP MCLIENTNGR LMANPEALKI LSAITQPMVV VAIVGLYRTG KSYLMNKLAG KKKGFSLGST VQSHTKGIWM WCVPHPKKP GHILVLLDTE GLGDVEKGDN QNDSWIFALA VLLSSTFVYN SIGTINQQAM DQLYYVTELT HRIRSKSSPD E NENEVEDS ADFVSFFPDF VWTLRDFSLD LEADGQPLTP DEYLTYSLKL KKGTSQKDET FNLPRLCIRK FFPKKKCFVF DR PVHRRKL AQLEKLQDEE LDPEFVQQVA DFCSYIFSNS KTKTLSGGIQ VNGPRLESLV LTYVNAISSG DLPCMENAVL ALA QIENSA AVQKAIAHYE QQMGQKVQLP TETLQELLDL HRDSEREAIE VFIRSSFKDV DHLFQKELAA QLEKKRDDFC KQNQ EASSD RCSALLQVIF SPLEEEVKAG IYSKPGGYRL FVQKLQDLKK KYYEEPRKGI QAEEILQTYL KSKESMTDAI LQTDQ TLTE KEKEIEVERV KAESAQASAK MLQEMQRKNE QMMEQKERSY QEHLKQLTEK MENDRVQLLK EQERTLALKL QEQEQL LKE GFQKESRIMK NEIQDLQTKM RRRKACTIS UniProtKB: Guanylate-binding protein 1 |
-分子 #2: GUANOSINE-5'-DIPHOSPHATE
| 分子 | 名称: GUANOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 2 / 式: GDP |
|---|---|
| 分子量 | 理論値: 443.201 Da |
| Chemical component information |  ChemComp-GDP: |
-分子 #3: ALUMINUM FLUORIDE
| 分子 | 名称: ALUMINUM FLUORIDE / タイプ: ligand / ID: 3 / コピー数: 2 / 式: AF3 |
|---|---|
| 分子量 | 理論値: 83.977 Da |
| Chemical component information |  ChemComp-AF3: |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.7 mg/mL | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
詳細: 5u mM HEPES pH7.4, 150 mM NaCl, 1 mM DTT, 200 uM GDP, 10 mM NaF, 300 uM AlCl3, 5 mM MgCl2 | ||||||||||||||||||||||||
| グリッド | モデル: Quantifoil / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE | ||||||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 99 % / チャンバー内温度: 22 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 105000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 105000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 2 / 実像数: 5214 / 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 空間: RECIPROCAL / プロトコル: OTHER 当てはまり具合の基準: Real-space cross correlation | ||||||||
| 得られたモデル |  PDB-8cqb: |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X