+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Carin 1 bacteriophage tail, connector and tail fibers assembly | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 | Bacteriophage sp. Cobetia Marina Virus Carin1 != Bacteriophage sp. Bacteriophage sp. Cobetia Marina Virus Carin1
| |||||||||
| 生物種 |   Bacteriophage sp. (ファージ) Bacteriophage sp. (ファージ) | |||||||||
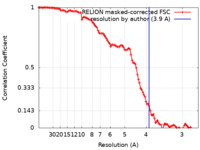
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.9 Å クライオ電子顕微鏡法 / 解像度: 3.9 Å | |||||||||
 データ登録者 データ登録者 | d'Acapito A / Neumann E / Schoehn G | |||||||||
| 資金援助 |  フランス, 1件 フランス, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Virol / 年: 2023 ジャーナル: J Virol / 年: 2023タイトル: Structural Study of the Cobetia marina Bacteriophage 1 (Carin-1) by Cryo-EM. 著者: Alessio d'Acapito / Thomas Roret / Eleftherios Zarkadas / Pierre-Yves Mocaër / Florian Lelchat / Anne-Claire Baudoux / Guy Schoehn / Emmanuelle Neumann /  要旨: Most of studied bacteriophages (phages) are terrestrial viruses. However, marine phages are shown to be highly involved in all levels of oceanic regulation. They are, however, still largely ...Most of studied bacteriophages (phages) are terrestrial viruses. However, marine phages are shown to be highly involved in all levels of oceanic regulation. They are, however, still largely overlooked by the scientific community. By inducing cell lysis on half of the bacterial population daily, their role and influence on the bacterial biomass and evolution, as well as their impact in the global biogeochemical cycles, is undeniable. Cobetia marina (Carin-1) is a member of the family infecting the γ. marina. Here, we present the almost complete, nearly-atomic resolution structure of Carin-1 comprising capsid, portal, and tail machineries at 3.5 Å, 3.8 Å and 3.9 Å, respectively, determined by cryo-electron microscopy (cryo-EM). Our experimental results, combined with AlphaFold2 (AF), allowed us to obtain the nearly-atomic structure of Carin-1 by fitting and refining the AF atomic models in the high resolution cryo-EM map, skipping the bottleneck of manual building and speeding up the structure determination process. Our structural results highlighted the T7-like nature of Carin1, as well as several novel structural features like the presence of short spikes on the capsid, reminiscent those described for Rhodobacter capsulatus gene transfer agent (RcGTA). This is, to our knowledge, the first time such assembly is described for a bacteriophage, shedding light into the common evolution and shared mechanisms between gene transfer agents and phages. This first full structure determined for a marine podophage allowed to propose an infection mechanism different than the one proposed for the archetypal podophage T7. Oceans play a central role in the carbon cycle on Earth and on the climate regulation (half of the planet's CO2 is absorbed by phytoplankton photosynthesis in the oceans and just as much O2 is liberated). The understanding of the biochemical equilibriums of marine biology represents a major goal for our future. By lysing half of the bacterial population every day, marine bacteriophages are key actors of these equilibriums. Despite their importance, these marine phages have, so far, only been studied a little and, in particular, structural insights are currently lacking, even though they are fundamental for the understanding of the molecular mechanisms of their mode of infection. The structures described in our manuscript allow us to propose an infection mechanism that differs from the one proposed for the terrestrial T7 virus, and might also allow us to, in the future, better understand the way bacteriophages shape the global ecosystem. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16689.map.gz emd_16689.map.gz | 94.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16689-v30.xml emd-16689-v30.xml emd-16689.xml emd-16689.xml | 18.2 KB 18.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16689_fsc.xml emd_16689_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16689.png emd_16689.png | 52.5 KB | ||
| その他 |  emd_16689_half_map_1.map.gz emd_16689_half_map_1.map.gz emd_16689_half_map_2.map.gz emd_16689_half_map_2.map.gz | 79.1 MB 79.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16689 http://ftp.pdbj.org/pub/emdb/structures/EMD-16689 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16689 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16689 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16689.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16689.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.35 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_16689_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
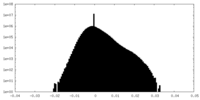
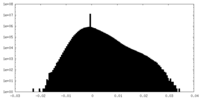
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_16689_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
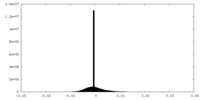
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Bacteriophage sp. Cobetia Marina Virus Carin1
| 全体 | 名称: Bacteriophage sp. Cobetia Marina Virus Carin1 |
|---|---|
| 要素 |
|
-超分子 #1: Bacteriophage sp.
| 超分子 | 名称: Bacteriophage sp. / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Cobetia Marina Virus Carin1 / NCBI-ID: 3801 / 生物種: Bacteriophage sp. / ウイルスタイプ: VIRION / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Cobetia marina (バクテリア) / 株: DSM 4741 Cobetia marina (バクテリア) / 株: DSM 4741 |
-分子 #1: Tail Nozzle
| 分子 | 名称: Tail Nozzle / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Bacteriophage sp. (ファージ) Bacteriophage sp. (ファージ) |
| 分子量 | 理論値: 91.324289 KDa |
| 配列 | 文字列: MASNNYQPAS SYIQPSFAGG ELAPSLQGRV DLARYAISLK TCRNFVVQPY GGASNRPGFR FNTACKYKNY ATRLIPFSFN TEQTYVIEI GHQYMRFHRD GAPVLDGGEP VEVATSWHRD DIFEIKYVQS ADVLTLVHPD YKPRQLKRYS ETDWVLDFFD N EFGPLQDQ ...文字列: MASNNYQPAS SYIQPSFAGG ELAPSLQGRV DLARYAISLK TCRNFVVQPY GGASNRPGFR FNTACKYKNY ATRLIPFSFN TEQTYVIEI GHQYMRFHRD GAPVLDGGEP VEVATSWHRD DIFEIKYVQS ADVLTLVHPD YKPRQLKRYS ETDWVLDFFD N EFGPLQDQ NVDESITIIS NGVVDLVELT ASEAIFSEAM VGTTIKLQQV SSGEVAAWQN RSAVEQGDLA YVDERTYKAT SL SGGVDNT LTGDNTPAHT EGEQWDGPRT TIQGVTETLG VKWAYLHSGF GYVRITEHRD DTHIVGRVIG RLPEEIRTEG TYR WSFAAW DSDRGYPGTA SYYQQRLVFA NSRAEPQAFW MSETGIFNGF KVSFPIEADD AITFTLASRQ VNEIRHLIPL GSLL ALTSG AEWMISDNDQ GLAPDTVSAD VQGYRGASDV TPLLIGSSAL YVQARGTVIR DLAYSFELDG YTGDDLTIFS NHLLK DYTI KDWAYAQEPD SVVWLVRSDG ALLSMTYQRE QQVVAWARHD TVDGEFESVA VIAEGSRDVP YAIVKRQVGG ETVRYI EYL DSRRFSHVED FFCVDSGLTY DGRSSTGALL TIGGGTNWTT DEDLTLTASA SSFSPSDVGR RVRVYTGDKF ADVDVDA YV SATSVAVSAV RIVPEELRGV QGDRWGFMAK TLTGLDHLEG KTVSILADGN VHAPEVVTGG QVTLDYSAAV VHVGLPIE S DIETLPISSS GATVRDSHKA IVGVGIQLEK SRGVFAARSR RDFTSSDLIE LKQRDAEDWG EATGLETGLV ELGIPTSWD KDGSLFIRQS DPLPLTILSI IPRVVMGGKG |
-分子 #2: Tail fibers Dpo36
| 分子 | 名称: Tail fibers Dpo36 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Bacteriophage sp. (ファージ) Bacteriophage sp. (ファージ) |
| 分子量 | 理論値: 86.858156 KDa |
| 配列 | 文字列: MTVPTNDNRE QYAGNGATTV FPYAFRIFES SDLEVYLTDE DGDQALLIEG TDYTVSGAGD EEGGEITFPV SGDPLDDGET LTILRVIDI TQETDLKNQG AYYPEVVEDE FDRSRMIDQQ QQEQLDRALI KTETGDRWEG QGVPAKNFAM SDPVEDTDLP T VRWTKDYV ...文字列: MTVPTNDNRE QYAGNGATTV FPYAFRIFES SDLEVYLTDE DGDQALLIEG TDYTVSGAGD EEGGEITFPV SGDPLDDGET LTILRVIDI TQETDLKNQG AYYPEVVEDE FDRSRMIDQQ QQEQLDRALI KTETGDRWEG QGVPAKNFAM SDPVEDTDLP T VRWTKDYV TQMAEGITGD IGAYTVVAPT SGDEKRLDEW MDDIQRPDDS LVVADGGTEA RSLSERFADS ASYQDYGIAG DG TTNDTAA FAALESDRSS DAIELHGNTY LVDEIPNGNA YRDAVWSLDG EDLSISEYGG LVTGTPTTGA FEPAYTGGVN NTP TTSGRT NKHTRAILAS QNCRADFARS ACVASIYSWA YGNVSGNFAS RQSIAGAPQT VNIGSEEGQA LGFQSGNYTT QFCR AEGST TFNIGSDDCA ASGAHSGTIS SLESYAGRGH DFRGTPVFDD GVLVDITIDD AGAGYVPGSD VMYLQNRQFG NTTDA VITY TVDGTGGVSA ITITDGGSGY SGIVAARIDT FGDYSLVMAS ARSKIEDQFC AAIASDNARV RGRESAVIAS DGGVVN EDN SVVIGSVDST SNGARSGIYT GSGCETTGAG AVVIGGVNAK ASNDGAIVMG RGVDSEFARS LVFGDGGSGA AASTAGR KF QVTAAGNVTA AGTITGSTTY ADYAEYFENS ARGVIPLGVI VTLDGRKVRP ASAGDDIIGV VSGTAILAAG DSQFHWGG R YLAGEFGELL YHDVDVDGKI ERQPVENPEY DPSVPNVPRS QRPEEWSCIG LVGQLHVRVS SDVAAGDRVA AGDGGIGVP GDNGMICMEI KQAYDSGKGY AVALCLHK |
-分子 #3: Connector Protein
| 分子 | 名称: Connector Protein / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Bacteriophage sp. (ファージ) Bacteriophage sp. (ファージ) |
| 分子量 | 理論値: 24.697715 KDa |
| 配列 | 文字列: MPSKVDICNR ALSNTGTDIT IASLTEKSKE ARLCQQWYDA TLASLLRTYQ WAFAQRRVTL ALIGVGPAGW RHKYRYPTDA ITIHDVFTA DTYPDGASEF TDGRYRQIFQ IASDGEGGRL VLANCEDAMC RYTSDIEDPN LMPPDFSTAL EMMLAKNIAM P MTGNPGLM ...文字列: MPSKVDICNR ALSNTGTDIT IASLTEKSKE ARLCQQWYDA TLASLLRTYQ WAFAQRRVTL ALIGVGPAGW RHKYRYPTDA ITIHDVFTA DTYPDGASEF TDGRYRQIFQ IASDGEGGRL VLANCEDAMC RYTSDIEDPN LMPPDFSTAL EMMLAKNIAM P MTGNPGLM TVLAQQAASL VSDAIARDQN EGYRNPLPYA SWTRANIGDS YPDDDHLPHR GGRR |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 / 構成要素 - 濃度: 1.0 x / 構成要素 - 名称: PBS |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER/RHODIUM / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. / 詳細: 25mA |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 4.0 µm Bright-field microscopy / 最大 デフォーカス(公称値): 4.0 µm最小 デフォーカス(公称値): 0.7000000000000001 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 30.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z
Z Y
Y X
X