+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | AQP7 dimer of tetramers_D4 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Transport of glycerol from adipocytes to the liver by Aquaporins / glycerol channel activity / urea transmembrane transporter activity / Passive transport by Aquaporins / glycerol transmembrane transport /  water channel activity / water transport / water channel activity / water transport /  lipid droplet / cytoplasmic vesicle membrane / cell-cell junction ...Transport of glycerol from adipocytes to the liver by Aquaporins / glycerol channel activity / urea transmembrane transporter activity / Passive transport by Aquaporins / glycerol transmembrane transport / lipid droplet / cytoplasmic vesicle membrane / cell-cell junction ...Transport of glycerol from adipocytes to the liver by Aquaporins / glycerol channel activity / urea transmembrane transporter activity / Passive transport by Aquaporins / glycerol transmembrane transport /  water channel activity / water transport / water channel activity / water transport /  lipid droplet / cytoplasmic vesicle membrane / cell-cell junction / lipid droplet / cytoplasmic vesicle membrane / cell-cell junction /  細胞皮質 / basolateral plasma membrane / 細胞皮質 / basolateral plasma membrane /  細胞膜 / 細胞膜 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.55 Å クライオ電子顕微鏡法 / 解像度: 2.55 Å | |||||||||
 データ登録者 データ登録者 | Huang P / Venskutonyte R / Fan X / Li P / Yan N / Gourdon P / Lindkvist-Petersson K | |||||||||
| 資金援助 |  スウェーデン, 2件 スウェーデン, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Cryo-EM structure supports a role of AQP7 as a junction protein. 著者: Peng Huang / Raminta Venskutonytė / Rashmi B Prasad / Hamidreza Ardalani / Sofia W de Maré / Xiao Fan / Ping Li / Peter Spégel / Nieng Yan / Pontus Gourdon / Isabella Artner / Karin Lindkvist-Petersson /   要旨: Aquaglyceroporin 7 (AQP7) facilitates glycerol flux across the plasma membrane with a critical physiological role linked to metabolism, obesity, and associated diseases. Here, we present the single- ...Aquaglyceroporin 7 (AQP7) facilitates glycerol flux across the plasma membrane with a critical physiological role linked to metabolism, obesity, and associated diseases. Here, we present the single-particle cryo-EM structure of AQP7 determined at 2.55 Å resolution adopting two adhering tetramers, stabilized by extracellularly exposed loops, in a configuration like that of the well-characterized interaction of AQP0 tetramers. The central pore, in-between the four monomers, displays well-defined densities restricted by two leucine filters. Gas chromatography mass spectrometry (GC/MS) results show that the AQP7 sample contains glycerol 3-phosphate (Gro3P), which is compatible with the identified features in the central pore. AQP7 is shown to be highly expressed in human pancreatic α- and β- cells suggesting that the identified AQP7 octamer assembly, in addition to its function as glycerol channel, may serve as junction proteins within the endocrine pancreas. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15528.map.gz emd_15528.map.gz | 125.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15528-v30.xml emd-15528-v30.xml emd-15528.xml emd-15528.xml | 17.6 KB 17.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_15528.png emd_15528.png | 38 KB | ||
| その他 |  emd_15528_half_map_1.map.gz emd_15528_half_map_1.map.gz emd_15528_half_map_2.map.gz emd_15528_half_map_2.map.gz | 225.9 MB 225.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15528 http://ftp.pdbj.org/pub/emdb/structures/EMD-15528 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15528 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15528 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15528.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15528.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8464 Å | ||||||||||||||||||||||||||||||||||||
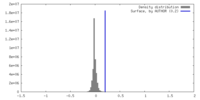
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_15528_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
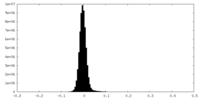
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_15528_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
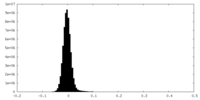
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : dimer of tetramers
| 全体 | 名称: dimer of tetramers |
|---|---|
| 要素 |
|
-超分子 #1: dimer of tetramers
| 超分子 | 名称: dimer of tetramers / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Aquaporin-7
| 分子 | 名称: Aquaporin-7 / タイプ: protein_or_peptide / ID: 1 / コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 37.264395 KDa |
| 組換発現 | 生物種:   Komagataella pastoris (菌類) Komagataella pastoris (菌類) |
| 配列 | 文字列: MVQASGHRRS TRGSKMVSWS VIAKIQEILQ RKMVREFLAE FMSTYVMMVF GLGSVAHMVL NKKYGSYLGV NLGFGFGVTM GVHVAGRIS GAHMNAAVTF ANCALGRVPW RKFPVYVLGQ FLGSFLAAAT IYSLFYTAIL HFSGGQLMVT GPVATAGIFA T YLPDHMTL ...文字列: MVQASGHRRS TRGSKMVSWS VIAKIQEILQ RKMVREFLAE FMSTYVMMVF GLGSVAHMVL NKKYGSYLGV NLGFGFGVTM GVHVAGRIS GAHMNAAVTF ANCALGRVPW RKFPVYVLGQ FLGSFLAAAT IYSLFYTAIL HFSGGQLMVT GPVATAGIFA T YLPDHMTL WRGFLNEAWL TGMLQLCLFA ITDQENNPAL PGTEALVIGI LVVIIGVSLG MNTGYAINPS RDLPPRIFTF IA GWGKQVF SNGENWWWVP VVAPLLGAYL GGIIYLVFIG STIPREPLKL EDSVAYEDHG ITVLPKMGSH EPTISPLTPV SVS PANRSS VHPAPPLHES MALEHF |
-分子 #2: water
| 分子 | 名称: water / タイプ: ligand / ID: 2 / コピー数: 57 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 詳細: 20mA | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK II / 詳細: 3s blot, 3s wait, 0s drain time, 0 blot force. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 105000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 105000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 14000 / 平均露光時間: 2.0 sec. / 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 371567 詳細: Those particles were picked from the sub-dataset of 3225 micrographs by template picking tool. |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 3次元分類 | クラス数: 5 / ソフトウェア - 名称: cryoSPARC |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 使用したクラス数: 1 想定した対称性 - 点群: D4 (2回x4回 2面回転対称  ) )解像度のタイプ: BY AUTHOR / 解像度: 2.55 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC / 使用した粒子像数: 102367 |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 温度因子: 98.6 |
|---|---|
| 得られたモデル |  PDB-8amx: |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)