+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10895 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the flagellar MotAB stator complex from Clostridium sporogenes | |||||||||||||||
 マップデータ マップデータ | post processed volume MotAB complex clostridium sporogenes | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Stator Flagellar rotation ion driven motor /  MOTOR PROTEIN (モータータンパク質) MOTOR PROTEIN (モータータンパク質) | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||||||||
| 生物種 |   Clostridium sporogenes (バクテリア) Clostridium sporogenes (バクテリア) | |||||||||||||||
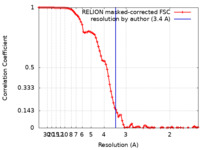
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.4 Å クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||||||||
 データ登録者 データ登録者 | Lea SM / Deme JC | |||||||||||||||
| 資金援助 |  英国, 4件 英国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Microbiol / 年: 2020 ジャーナル: Nat Microbiol / 年: 2020タイトル: Structures of the stator complex that drives rotation of the bacterial flagellum. 著者: Justin C Deme / Steven Johnson / Owen Vickery / Amy Aron / Holly Monkhouse / Thomas Griffiths / Rory Hennell James / Ben C Berks / James W Coulton / Phillip J Stansfeld / Susan M Lea /   要旨: The bacterial flagellum is the prototypical protein nanomachine and comprises a rotating helical propeller attached to a membrane-embedded motor complex. The motor consists of a central rotor ...The bacterial flagellum is the prototypical protein nanomachine and comprises a rotating helical propeller attached to a membrane-embedded motor complex. The motor consists of a central rotor surrounded by stator units that couple ion flow across the cytoplasmic membrane to generate torque. Here, we present the structures of the stator complexes from Clostridium sporogenes, Bacillus subtilis and Vibrio mimicus, allowing interpretation of the extensive body of data on stator mechanism. The structures reveal an unexpected asymmetric AB subunit assembly where the five A subunits enclose the two B subunits. Comparison to structures of other ion-driven motors indicates that this AB architecture is fundamental to bacterial systems that couple energy from ion flow to generate mechanical work at a distance and suggests that such events involve rotation in the motor structures. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10895.map.gz emd_10895.map.gz | 59.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10895-v30.xml emd-10895-v30.xml emd-10895.xml emd-10895.xml | 22.1 KB 22.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10895_fsc.xml emd_10895_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10895.png emd_10895.png | 235.3 KB | ||
| マスクデータ |  emd_10895_msk_1.map emd_10895_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-10895.cif.gz emd-10895.cif.gz | 5.9 KB | ||
| その他 |  emd_10895_additional_1.map.gz emd_10895_additional_1.map.gz emd_10895_additional_2.map.gz emd_10895_additional_2.map.gz emd_10895_half_map_1.map.gz emd_10895_half_map_1.map.gz emd_10895_half_map_2.map.gz emd_10895_half_map_2.map.gz | 41.3 MB 49.1 MB 49.6 MB 49.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10895 http://ftp.pdbj.org/pub/emdb/structures/EMD-10895 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10895 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10895 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10895.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10895.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | post processed volume MotAB complex clostridium sporogenes | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.822 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10895_msk_1.map emd_10895_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
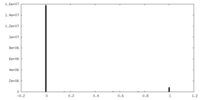
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Relion Local Resolution filtered volume MotAB clostridium sporogenes
| ファイル | emd_10895_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Relion Local Resolution filtered volume MotAB clostridium sporogenes | ||||||||||||
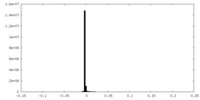
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Refinement volume MotAB clostridium sporogenes
| ファイル | emd_10895_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Refinement volume MotAB clostridium sporogenes | ||||||||||||
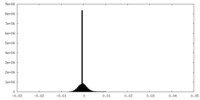
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_10895_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
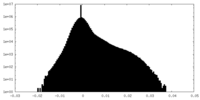
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_10895_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : MotAB
| 全体 | 名称: MotAB |
|---|---|
| 要素 |
|
-超分子 #1: MotAB
| 超分子 | 名称: MotAB / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Clostridium sporogenes (バクテリア) Clostridium sporogenes (バクテリア) |
-分子 #1: Chemotaxis motB protein
| 分子 | 名称: Chemotaxis motB protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Clostridium sporogenes (バクテリア) Clostridium sporogenes (バクテリア) |
| 分子量 | 理論値: 27.948547 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MARRNKKGGG GDEIRGDEWL ATFSDTITLL LTFFILLYSF SSVDAQKFQQ VASAMQVAMT GQSGNTIVDY NLKNGDVPLV GETTKLGRE TGSDAKSDSK EVYNEVNKFV DKNNLKSSVE VKEDGRGVII QLRDNVLFEI GRADIKPQSK QIMDKINGLI A TLPNEVIV ...文字列: MARRNKKGGG GDEIRGDEWL ATFSDTITLL LTFFILLYSF SSVDAQKFQQ VASAMQVAMT GQSGNTIVDY NLKNGDVPLV GETTKLGRE TGSDAKSDSK EVYNEVNKFV DKNNLKSSVE VKEDGRGVII QLRDNVLFEI GRADIKPQSK QIMDKINGLI A TLPNEVIV EGHTDNVPIK NEVYGSNWEL STARAVNVLR YFVETKKQNP VRFTAAGYGE YRPIAQNNSD VNKAKNRRVN IV IVSKEKE SSKK UniProtKB:  Chemotaxis protein MotB Chemotaxis protein MotB |
-分子 #2: Chemotaxis MotA protein
| 分子 | 名称: Chemotaxis MotA protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Clostridium sporogenes (バクテリア) Clostridium sporogenes (バクテリア) |
| 分子量 | 理論値: 29.613506 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MKKRDILTPI GFVLCFGLVL WGMASGGSNL KVFWDVASVF ITIGGSMAAM LITYPMDEFK RLLIVIRQTF KDNGMSNIDV IQNFVDLSR KARREGLLSL EDAINNLTDD YMKKGLRMVV DGIEPETIRE IMELEIDEME KRHKSGADML KTWGGYAPAF G MVGTLIGL ...文字列: MKKRDILTPI GFVLCFGLVL WGMASGGSNL KVFWDVASVF ITIGGSMAAM LITYPMDEFK RLLIVIRQTF KDNGMSNIDV IQNFVDLSR KARREGLLSL EDAINNLTDD YMKKGLRMVV DGIEPETIRE IMELEIDEME KRHKSGADML KTWGGYAPAF G MVGTLIGL IQMLANLTDS STIASGMGKA LITTFYGSLM ANAVFNPMGA NLMFKSGVEA TTREMVLEGV LAIQSGVNPR IM EEKLVSY LSPPERQAYS KVQVSGEGAA QNG UniProtKB: Chemotaxis MotA protein |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 0.0003 µm / 最小 デフォーカス(公称値): 0.0001 µm / 倍率(公称値): 165000 Bright-field microscopy / 最大 デフォーカス(公称値): 0.0003 µm / 最小 デフォーカス(公称値): 0.0001 µm / 倍率(公称値): 165000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 48.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-6ysf: |
 ムービー
ムービー コントローラー
コントローラー



















 Z
Z Y
Y X
X