+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0834 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the peripheral FCPI from diatom | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Chaetoceros gracilis (珪藻) Chaetoceros gracilis (珪藻) | |||||||||
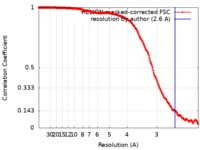
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.6 Å クライオ電子顕微鏡法 / 解像度: 2.6 Å | |||||||||
 データ登録者 データ登録者 | Nagao R / Kato K / Miyazaki N / Akita F / Shen JR | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: Structural basis for assembly and function of a diatom photosystem I-light-harvesting supercomplex. 著者: Ryo Nagao / Koji Kato / Kentaro Ifuku / Takehiro Suzuki / Minoru Kumazawa / Ikuo Uchiyama / Yasuhiro Kashino / Naoshi Dohmae / Seiji Akimoto / Jian-Ren Shen / Naoyuki Miyazaki / Fusamichi Akita /  要旨: Photosynthetic light-harvesting complexes (LHCs) play a pivotal role in collecting solar energy for photochemical reactions in photosynthesis. One of the major LHCs are fucoxanthin chlorophyll a/c- ...Photosynthetic light-harvesting complexes (LHCs) play a pivotal role in collecting solar energy for photochemical reactions in photosynthesis. One of the major LHCs are fucoxanthin chlorophyll a/c-binding proteins (FCPs) present in diatoms, a group of organisms having important contribution to the global carbon cycle. Here, we report a 2.40-Å resolution structure of the diatom photosystem I (PSI)-FCPI supercomplex by cryo-electron microscopy. The supercomplex is composed of 16 different FCPI subunits surrounding a monomeric PSI core. Each FCPI subunit showed different protein structures with different pigment contents and binding sites, and they form a complicated pigment-protein network together with the PSI core to harvest and transfer the light energy efficiently. In addition, two unique, previously unidentified subunits were found in the PSI core. The structure provides numerous insights into not only the light-harvesting strategy in diatom PSI-FCPI but also evolutionary dynamics of light harvesters among oxyphototrophs. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0834.map.gz emd_0834.map.gz | 28.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0834-v30.xml emd-0834-v30.xml emd-0834.xml emd-0834.xml | 23.8 KB 23.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0834_fsc.xml emd_0834_fsc.xml | 17.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0834.png emd_0834.png | 77.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0834 http://ftp.pdbj.org/pub/emdb/structures/EMD-0834 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0834 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0834 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0834.map.gz / 形式: CCP4 / 大きさ: 488.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0834.map.gz / 形式: CCP4 / 大きさ: 488.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.113 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : peripheral FCPI
+超分子 #1: peripheral FCPI
+分子 #1: Fucoxanthin chlorophyll a/c-binding protein Lhcr12
+分子 #2: Fucoxanthin chlorophyll a/c-binding protein Lhcr10
+分子 #3: Fucoxanthin chlorophyll a/c-binding protein Lhcr4
+分子 #4: Fucoxanthin chlorophyll a/c-binding protein Lhcr3
+分子 #5: Fucoxanthin chlorophyll a/c-binding protein Lhcq13
+分子 #6: Fucoxanthin chlorophyll a/c-binding protein Lhcq3
+分子 #7: Fucoxanthin chlorophyll a/c-binding protein Lhcq11
+分子 #8: Fucoxanthin chlorophyll a/c-binding protein Lhcq10
+分子 #9: Fucoxanthin chlorophyll a/c-binding protein Lhcq8
+分子 #10: Fucoxanthin chlorophyll a/c-binding protein Lhcq5
+分子 #11: CHLOROPHYLL A
+分子 #12: Chlorophyll c1
+分子 #13: (3S,3'R,5R,6S,7cis)-7',8'-didehydro-5,6-dihydro-5,6-epoxy-beta,be...
+分子 #14: (3S,3'S,5R,5'R,6S,6'R,8'R)-3,5'-dihydroxy-8-oxo-6',7'-didehydro-5...
+分子 #15: 1,2-DIPALMITOYL-PHOSPHATIDYL-GLYCEROLE
+分子 #16: 1,2-DISTEAROYL-MONOGALACTOSYL-DIGLYCERIDE
+分子 #17: DODECYL-BETA-D-MALTOSIDE
+分子 #18: water
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 6.5 構成要素:
| |||||||||
| グリッド | モデル: C-flat-1.2/1.3 / 材質: MOLYBDENUM / メッシュ: 300 / 支持フィルム - 材質: CARBON / 前処理 - タイプ: PLASMA CLEANING | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-6l4t: |
 ムービー
ムービー コントローラー
コントローラー