+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0697 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 200kV MicroED structure of FUS (37-42) SYSGYS solved from single crystal at 0.67 A | |||||||||||||||
 マップデータ マップデータ | 2Fo-Fc map | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | FUS /  MicroED / Ultrahigh resolution / MicroED / Ultrahigh resolution /  RNA BINDING PROTEIN (RNA結合タンパク質) RNA BINDING PROTEIN (RNA結合タンパク質) | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mRNA stabilization / intracellular non-membrane-bounded organelle /  regulation of RNA splicing / Processing of Capped Intron-Containing Pre-mRNA / positive regulation of double-strand break repair via homologous recombination / mRNA Splicing - Major Pathway / regulation of RNA splicing / Processing of Capped Intron-Containing Pre-mRNA / positive regulation of double-strand break repair via homologous recombination / mRNA Splicing - Major Pathway /  RNA splicing / molecular condensate scaffold activity / mRNA 3'-UTR binding / RNA splicing / molecular condensate scaffold activity / mRNA 3'-UTR binding /  transcription coregulator activity ...mRNA stabilization / intracellular non-membrane-bounded organelle / transcription coregulator activity ...mRNA stabilization / intracellular non-membrane-bounded organelle /  regulation of RNA splicing / Processing of Capped Intron-Containing Pre-mRNA / positive regulation of double-strand break repair via homologous recombination / mRNA Splicing - Major Pathway / regulation of RNA splicing / Processing of Capped Intron-Containing Pre-mRNA / positive regulation of double-strand break repair via homologous recombination / mRNA Splicing - Major Pathway /  RNA splicing / molecular condensate scaffold activity / mRNA 3'-UTR binding / RNA splicing / molecular condensate scaffold activity / mRNA 3'-UTR binding /  transcription coregulator activity / protein homooligomerization / amyloid fibril formation / transcription coregulator activity / protein homooligomerization / amyloid fibril formation /  transcription coactivator activity / transcription coactivator activity /  chromatin binding / regulation of DNA-templated transcription / regulation of transcription by RNA polymerase II / chromatin binding / regulation of DNA-templated transcription / regulation of transcription by RNA polymerase II /  DNA binding / DNA binding /  RNA binding / RNA binding /  核質 / identical protein binding / 核質 / identical protein binding /  metal ion binding / metal ion binding /  細胞核 / 細胞核 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||
| 手法 |  電子線結晶学 / 電子線結晶学 /  クライオ電子顕微鏡法 / 解像度: 0.67 Å クライオ電子顕微鏡法 / 解像度: 0.67 Å | |||||||||||||||
 データ登録者 データ登録者 | Zhou H / Luo F | |||||||||||||||
| 資金援助 |  中国, 4件 中国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Anal Chem / 年: 2019 ジャーナル: Anal Chem / 年: 2019タイトル: Programming Conventional Electron Microscopes for Solving Ultrahigh-Resolution Structures of Small and Macro-Molecules. 著者: Heng Zhou / Feng Luo / Zhipu Luo / Dan Li / Cong Liu / Xueming Li /  要旨: Microcrystal electron diffraction (MicroED) is becoming a powerful tool in determining the crystal structures of biological macromolecules and small organic compounds. However, wide applications of ...Microcrystal electron diffraction (MicroED) is becoming a powerful tool in determining the crystal structures of biological macromolecules and small organic compounds. However, wide applications of this technique are still limited by the special requirement for radiation-tolerated movie-mode camera and the lack of automated data collection methods. Herein, we develop a stage-camera synchronization scheme to minimize the hardware requirements and enable the use of the conventional electron cryo-microscope with a single-frame CCD camera, which ensures not only the acquisition of ultrahigh-resolution diffraction data but also low cost in practice. This method renders the structure determination of both peptide and small organic compounds at ultrahigh resolution up to ∼0.60 Å with unambiguous assignment of nearly all hydrogen atoms. The present work provides a widely applicable solution for routine structure determination of MicroED and demonstrates the capability of the low-end 120 kV microscope with a CCD camera in solving ultrahigh resolution structures of both organic compounds and biological macromolecules. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0697.map.gz emd_0697.map.gz | 1.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0697-v30.xml emd-0697-v30.xml emd-0697.xml emd-0697.xml | 12.6 KB 12.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_0697.png emd_0697.png | 49.4 KB | ||
| Filedesc metadata |  emd-0697.cif.gz emd-0697.cif.gz | 4.2 KB | ||
| その他 |  emd_0697_additional.map.gz emd_0697_additional.map.gz emd_0697_additional_1.map.gz emd_0697_additional_1.map.gz | 1.7 MB 1.7 MB | ||
| Filedesc structureFactors |  emd_0697_sf.cif.gz emd_0697_sf.cif.gz | 124.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0697 http://ftp.pdbj.org/pub/emdb/structures/EMD-0697 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0697 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0697 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6kj2MC  0696C  0698C  0699C  6kj1C  6kj3C  6kj4C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0697.map.gz / 形式: CCP4 / 大きさ: 9.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0697.map.gz / 形式: CCP4 / 大きさ: 9.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 2Fo-Fc map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X: 0.15092 Å / Y: 0.16497 Å / Z: 0.15554 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Fo-Fc map
| ファイル | emd_0697_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Fo-Fc map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
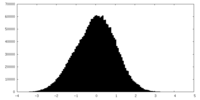
| 密度ヒストグラム |
-追加マップ: Fo-Fc map
| ファイル | emd_0697_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Fo-Fc map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
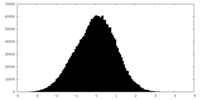
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : FUS LC RAC1
| 全体 | 名称: FUS LC RAC1 |
|---|---|
| 要素 |
|
-超分子 #1: FUS LC RAC1
| 超分子 | 名称: FUS LC RAC1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|
-分子 #1: RNA-binding protein FUS
| 分子 | 名称: RNA-binding protein FUS / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 662.648 Da |
| 配列 | 文字列: SYSGYS UniProtKB:  FUS FUS |
-分子 #2: water
| 分子 | 名称: water / タイプ: ligand / ID: 2 / コピー数: 1 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  電子線結晶学 電子線結晶学 |
| 試料の集合状態 | 3D array |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: DIFFRACTION 回折 / カメラ長: 520 mm 回折 / カメラ長: 520 mm |
| 撮影 | フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 平均電子線量: 0.05 e/Å2 |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| Crystallography statistics | Number intensities measured: 9323 / Number structure factors: 3780 / Fourier space coverage: 58.77 / R sym: 0.123 / R merge: 0.123 / Overall phase error: 40.93 / Overall phase residual: 40.93 / Phase error rejection criteria: 1 / High resolution: 0.67 Å / 殻 - Shell ID: 1 / 殻 - High resolution: 0.67 Å / 殻 - Low resolution: 0.69 Å / 殻 - Number structure factors: 227 / 殻 - Phase residual: 1 / 殻 - Fourier space coverage: 35.97 / 殻 - Multiplicity: 1.99 |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 0.67 Å / 解像度の算出法: DIFFRACTION PATTERN/LAYERLINES |
-原子モデル構築 1
| 精密化 | 空間: RECIPROCAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-6kj2: |
 ムービー
ムービー コントローラー
コントローラー








 Z
Z Y
Y X
X