+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0235 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of Influenza Hemagglutinin ectodomain (A/duck/Alberta/35/76) in complex with FISW84 Fab Fragment | ||||||||||||||||||||||||
 マップデータ マップデータ | Sharpened Map of A/duck/Alberta/35/76 Hemagglutinin Ectodomain in Complex with FISW84 Fab | ||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 viral budding from plasma membrane / clathrin-dependent endocytosis of virus by host cell / host cell surface receptor binding / apical plasma membrane / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral budding from plasma membrane / clathrin-dependent endocytosis of virus by host cell / host cell surface receptor binding / apical plasma membrane / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) / virion attachment to host cell / host cell plasma membrane / virion membrane / エンベロープ (ウイルス) / virion attachment to host cell / host cell plasma membrane / virion membrane /  生体膜 生体膜類似検索 - 分子機能 | ||||||||||||||||||||||||
| 生物種 |   Influenza A virus (strain A/Duck/Alberta/35/1976 H1N1) (A型インフルエンザウイルス) / Influenza A virus (strain A/Duck/Alberta/35/1976 H1N1) (A型インフルエンザウイルス) /   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.3 Å クライオ電子顕微鏡法 / 解像度: 3.3 Å | ||||||||||||||||||||||||
 データ登録者 データ登録者 | Benton DJ / Rosenthal PB | ||||||||||||||||||||||||
| 資金援助 |  英国, 7件 英国, 7件
| ||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2018 ジャーナル: Proc Natl Acad Sci U S A / 年: 2018タイトル: Influenza hemagglutinin membrane anchor. 著者: Donald J Benton / Andrea Nans / Lesley J Calder / Jack Turner / Ursula Neu / Yi Pu Lin / Esther Ketelaars / Nicole L Kallewaard / Davide Corti / Antonio Lanzavecchia / Steven J Gamblin / ...著者: Donald J Benton / Andrea Nans / Lesley J Calder / Jack Turner / Ursula Neu / Yi Pu Lin / Esther Ketelaars / Nicole L Kallewaard / Davide Corti / Antonio Lanzavecchia / Steven J Gamblin / Peter B Rosenthal / John J Skehel /    要旨: Viruses with membranes fuse them with cellular membranes, to transfer their genomes into cells at the beginning of infection. For Influenza virus, the membrane glycoprotein involved in fusion is the ...Viruses with membranes fuse them with cellular membranes, to transfer their genomes into cells at the beginning of infection. For Influenza virus, the membrane glycoprotein involved in fusion is the hemagglutinin (HA), the 3D structure of which is known from X-ray crystallographic studies. The soluble ectodomain fragments used in these studies lacked the "membrane anchor" portion of the molecule. Since this region has a role in membrane fusion, we have determined its structure by analyzing the intact, full-length molecule in a detergent micelle, using cryo-EM. We have also compared the structures of full-length HA-detergent micelles with full-length HA-Fab complex detergent micelles, to describe an infectivity-neutralizing monoclonal Fab that binds near the ectodomain membrane anchor junction. We determine a high-resolution HA structure which compares favorably in detail with the structure of the ectodomain seen by X-ray crystallography; we detect, clearly, all five carbohydrate side chains of HA; and we find that the ectodomain is joined to the membrane anchor by flexible, eight-residue-long, linkers. The linkers extend into the detergent micelle to join a central triple-helical structure that is a major component of the membrane anchor. | ||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0235.map.gz emd_0235.map.gz | 475.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0235-v30.xml emd-0235-v30.xml emd-0235.xml emd-0235.xml | 24.8 KB 24.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0235_fsc.xml emd_0235_fsc.xml | 18.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0235.png emd_0235.png | 51 KB | ||
| マスクデータ |  emd_0235_msk_1.map emd_0235_msk_1.map | 512 MB |  マスクマップ マスクマップ | |
| その他 |  emd_0235_additional.map.gz emd_0235_additional.map.gz emd_0235_half_map_1.map.gz emd_0235_half_map_1.map.gz emd_0235_half_map_2.map.gz emd_0235_half_map_2.map.gz | 475.7 MB 475 MB 475 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0235 http://ftp.pdbj.org/pub/emdb/structures/EMD-0235 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0235 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0235 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0235.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0235.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened Map of A/duck/Alberta/35/76 Hemagglutinin Ectodomain in Complex with FISW84 Fab | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.078 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_0235_msk_1.map emd_0235_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
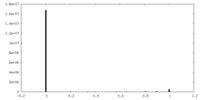
| 密度ヒストグラム |
-追加マップ: Unsharpened Map of A/duck/Alberta/35/76 Hemagglutinin Ectodomain in Complex...
| ファイル | emd_0235_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened Map of A/duck/Alberta/35/76 Hemagglutinin Ectodomain in Complex with FISW84 Fab | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
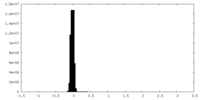
| 密度ヒストグラム |
-ハーフマップ: Half map 2 of A/duck/Alberta/35/76 Hemagglutinin Ectodomain in...
| ファイル | emd_0235_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 of A/duck/Alberta/35/76 Hemagglutinin Ectodomain in Complex with FISW84 Fab | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
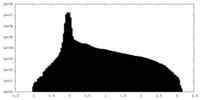
| 密度ヒストグラム |
-ハーフマップ: Half map 1 of A/duck/Alberta/35/76 Hemagglutinin Ectodomain in...
| ファイル | emd_0235_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 of A/duck/Alberta/35/76 Hemagglutinin Ectodomain in Complex with FISW84 Fab | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
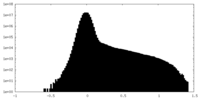
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of A/duck/Alberta/35/76 Haemagglutinin and FISW84 Fab Fragment
| 全体 | 名称: Complex of A/duck/Alberta/35/76 Haemagglutinin and FISW84 Fab Fragment |
|---|---|
| 要素 |
|
-超分子 #1: Complex of A/duck/Alberta/35/76 Haemagglutinin and FISW84 Fab Fragment
| 超分子 | 名称: Complex of A/duck/Alberta/35/76 Haemagglutinin and FISW84 Fab Fragment タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 分子量 | 理論値: 300 KDa |
-超分子 #2: A/duck/Alberta/35/76 Haemagglutinin
| 超分子 | 名称: A/duck/Alberta/35/76 Haemagglutinin / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Influenza A virus (strain A/Duck/Alberta/35/1976 H1N1) (A型インフルエンザウイルス) Influenza A virus (strain A/Duck/Alberta/35/1976 H1N1) (A型インフルエンザウイルス) |
-超分子 #3: FISW84 Fab Fragment
| 超分子 | 名称: FISW84 Fab Fragment / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #3-#4 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Hemagglutinin
| 分子 | 名称: Hemagglutinin / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Influenza A virus (strain A/Duck/Alberta/35/1976 H1N1) (A型インフルエンザウイルス) Influenza A virus (strain A/Duck/Alberta/35/1976 H1N1) (A型インフルエンザウイルス) |
| 分子量 | 理論値: 35.684852 KDa |
| 配列 | 文字列: DTICVGYHAN NSTDTVDTVL EKNVTVTHSV NLLEDSHNGK LCSLNGIAPL QLGKCNVAGW LLGNPECDLL LTANSWSYII ETSNSENGT CYPGEFIDYE ELREQLSSVS SFEKFEIFPK ASSWPNHETT KGVTAACSYS GASSFYRNLL WITKKGTSYP K LSKSYTNN ...文字列: DTICVGYHAN NSTDTVDTVL EKNVTVTHSV NLLEDSHNGK LCSLNGIAPL QLGKCNVAGW LLGNPECDLL LTANSWSYII ETSNSENGT CYPGEFIDYE ELREQLSSVS SFEKFEIFPK ASSWPNHETT KGVTAACSYS GASSFYRNLL WITKKGTSYP K LSKSYTNN KGKEVLVLWG VHHPPSVSEQ QSLYQNADAY VSVGSSKYNR RFAPEIAARP KVRGQAGRMN YYWTLLDQGD TI TFEATGN LIAPWYAFAL NKGSDSGIIT SDAPVHNCDT RCQTPHGALN SSLPFQNVHP ITIGECPKYV KSTKLRMATG LRN VPSI |
-分子 #2: Hemagglutinin
| 分子 | 名称: Hemagglutinin / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Influenza A virus (strain A/Duck/Alberta/35/1976 H1N1) (A型インフルエンザウイルス) Influenza A virus (strain A/Duck/Alberta/35/1976 H1N1) (A型インフルエンザウイルス) |
| 分子量 | 理論値: 19.953924 KDa |
| 配列 | 文字列: GLFGAIAGFI EGGWTGMIDG WYGYHHQNEQ GSGYAADQKS TQNAIDGITS KVNSVIEKMN TQFTAVGKEF NNLERRIENL NKKVDDGFL DVWTYNAELL VLLENERTLD FHDSNVRNLY EKVKSQLRNN AKEIGNGCFE FYHKCDDECM ESVKNGTYDY P KYSEESKL NREEID |
-分子 #3: Heavy chain of FISW84 Fab Fragment
| 分子 | 名称: Heavy chain of FISW84 Fab Fragment / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 22.925875 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: EVQLLESGGG LVQPGGSLRL SCAASGFTFS SYGMAWVRQA PGKGLEWVSF ISATGLSTYF ADSVKGRFTI SRDTTKNTLY LQMNSLRAD DTAVYFCARM RRTMIAFGGN DFWGQGTLVT VSSASTKGPS VFPLAPSGTA ALGCLVKDYF PEPVTVSWNS G ALTSGVHT ...文字列: EVQLLESGGG LVQPGGSLRL SCAASGFTFS SYGMAWVRQA PGKGLEWVSF ISATGLSTYF ADSVKGRFTI SRDTTKNTLY LQMNSLRAD DTAVYFCARM RRTMIAFGGN DFWGQGTLVT VSSASTKGPS VFPLAPSGTA ALGCLVKDYF PEPVTVSWNS G ALTSGVHT FPAVLQSSGL YSLSSVVTVP SSSLGTQTYI CNVNHKPSNT KVDKRVEP |
-分子 #4: Light chain of FISW84 Fab Fragment
| 分子 | 名称: Light chain of FISW84 Fab Fragment / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.277766 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: EVVMTQSPAT LSVSPGEGAT LSCRASQSVN TNVAWYQQKP GQAPRLLIYG ASTRATGIPA RFSGSGSGTE FTLTISTLQS EDFAVYYCQ QYSNWPPITF GQGTRLEIKR TVAAPSVFIF PPSDEQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN S QESVTEQD ...文字列: EVVMTQSPAT LSVSPGEGAT LSCRASQSVN TNVAWYQQKP GQAPRLLIYG ASTRATGIPA RFSGSGSGTE FTLTISTLQS EDFAVYYCQ QYSNWPPITF GQGTRLEIKR TVAAPSVFIF PPSDEQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN S QESVTEQD SKDSTYSLSS TLTLSKADYE KHKVYACEVT HQGLSSPVTK SFNRGE |
-分子 #9: UNKNOWN BRANCHED FRAGMENT OF PHOSPHOLIPID
| 分子 | 名称: UNKNOWN BRANCHED FRAGMENT OF PHOSPHOLIPID / タイプ: ligand / ID: 9 / コピー数: 3 / 式: UPL |
|---|---|
| 分子量 | 理論値: 478.92 Da |
| Chemical component information | 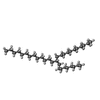 ChemComp-UPL: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.5 mg/mL | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||||
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm Bright-field microscopy / Cs: 2.7 mm |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 43.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー























 Z
Z Y
Y X
X