+検索条件
-Structure paper
| タイトル | Structural basis for receptor binding and broader interspecies receptor recognition of currently circulating Omicron sub-variants. |
|---|---|
| ジャーナル・号・ページ | Nat Commun, Vol. 14, Issue 1, Page 4405, Year 2023 |
| 掲載日 | 2023年7月21日 |
 著者 著者 | Zhennan Zhao / Yufeng Xie / Bin Bai / Chunliang Luo / Jingya Zhou / Weiwei Li / Yumin Meng / Linjie Li / Dedong Li / Xiaomei Li / Xiaoxiong Li / Xiaoyun Wang / Junqing Sun / Zepeng Xu / Yeping Sun / Wei Zhang / Zheng Fan / Xin Zhao / Linhuan Wu / Juncai Ma / Odel Y Li / Guijun Shang / Yan Chai / Kefang Liu / Peiyi Wang / George F Gao / Jianxun Qi /  |
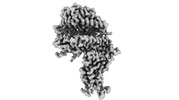
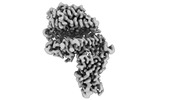
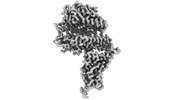
| PubMed 要旨 | Multiple SARS-CoV-2 Omicron sub-variants, such as BA.2, BA.2.12.1, BA.4, and BA.5, emerge one after another. BA.5 has become the dominant strain worldwide. Additionally, BA.2.75 is significantly ...Multiple SARS-CoV-2 Omicron sub-variants, such as BA.2, BA.2.12.1, BA.4, and BA.5, emerge one after another. BA.5 has become the dominant strain worldwide. Additionally, BA.2.75 is significantly increasing in some countries. Exploring their receptor binding and interspecies transmission risk is urgently needed. Herein, we examine the binding capacities of human and other 28 animal ACE2 orthologs covering nine orders towards S proteins of these sub-variants. The binding affinities between hACE2 and these sub-variants remain in the range as that of previous variants of concerns (VOCs) or interests (VOIs). Notably, R493Q reverse mutation enhances the bindings towards ACE2s from humans and many animals closely related to human life, suggesting an increased risk of cross-species transmission. Structures of S/hACE2 or RBD/hACE2 complexes for these sub-variants and BA.2 S binding to ACE2 of mouse, rat or golden hamster are determined to reveal the molecular basis for receptor binding and broader interspecies recognition. |
 リンク リンク |  Nat Commun / Nat Commun /  PubMed:37479708 / PubMed:37479708 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) / X線回折 |
| 解像度 | 2.58 - 3.29 Å |
| 構造データ | EMDB-33841, PDB-7yhw: EMDB-33870, PDB-7yj3: EMDB-34120, PDB-7yv8: EMDB-34138, PDB-7yvu: EMDB-34217, PDB-8gry: EMDB-34409, PDB-8h06:  EMDB-34494: Cryo-EM map of SARS-CoV-2 Omicron BA.2 spike trimer in complex with human ACE2 (three-RBD-up conformation)  EMDB-34498: Cryo-EM map of SARS-CoV-2 Omicron BA.2.12.1 spike trimer in complex with human ACE2 (three-RBD-up conformation)  EMDB-34499: Cryo-EM map of SARS-CoV-2 Omicron BA.2 spike protein in complex with mouse ACE2  EMDB-34506: Cryo-EM map of SARS-CoV-2 Omicron BA.2 spike trimer in complex with rat ACE2  EMDB-34509: Cryo-EM map of SARS-CoV-2 Omicron BA.4/5 (N658S) spike trimer in complex with human ACE2 (three-RBD-up conformation) 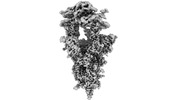 EMDB-34510: Cryo-EM map of SARS-CoV-2 Omicron BA.2 spike trimer in complex with golden hamster ACE2  PDB-8h5c: |
| 化合物 |  ChemComp-NAG:  ChemComp-ZN: |
| 由来 |
|
 キーワード キーワード | HYDROLASE/VIRAL PROTEIN /  SARS-CoV-2 (SARSコロナウイルス2) / Omicron BA.2.12.1 / SARS-CoV-2 (SARSコロナウイルス2) / Omicron BA.2.12.1 /  spike protein (スパイクタンパク質) / spike protein (スパイクタンパク質) /  VIRAL PROTEIN (ウイルスタンパク質) / HYDROLASE-VIRAL PROTEIN complex / Omicron BA.2 / Omicron BA.4/5 / Omicron BA.2.75 / VIRAL PROTEIN (ウイルスタンパク質) / HYDROLASE-VIRAL PROTEIN complex / Omicron BA.2 / Omicron BA.4/5 / Omicron BA.2.75 /  hACE2 (アンジオテンシン変換酵素2) hACE2 (アンジオテンシン変換酵素2) |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について