+検索条件
-Structure paper
| タイトル | Insights into distinct signaling profiles of the µOR activated by diverse agonists. |
|---|---|
| ジャーナル・号・ページ | Nat Chem Biol, Vol. 19, Issue 4, Page 423-430, Year 2023 |
| 掲載日 | 2022年11月21日 |
 著者 著者 | Qianhui Qu / Weijiao Huang / Deniz Aydin / Joseph M Paggi / Alpay B Seven / Haoqing Wang / Soumen Chakraborty / Tao Che / Jeffrey F DiBerto / Michael J Robertson / Asuka Inoue / Carl-Mikael Suomivuori / Bryan L Roth / Susruta Majumdar / Ron O Dror / Brian K Kobilka / Georgios Skiniotis /    |
| PubMed 要旨 | Drugs targeting the μ-opioid receptor (μOR) are the most effective analgesics available but are also associated with fatal respiratory depression through a pathway that remains unclear. Here we ...Drugs targeting the μ-opioid receptor (μOR) are the most effective analgesics available but are also associated with fatal respiratory depression through a pathway that remains unclear. Here we investigated the mechanistic basis of action of lofentanil (LFT) and mitragynine pseudoindoxyl (MP), two μOR agonists with different safety profiles. LFT, one of the most lethal opioids, and MP, a kratom plant derivative with reduced respiratory depression in animal studies, exhibited markedly different efficacy profiles for G protein subtype activation and β-arrestin recruitment. Cryo-EM structures of μOR-Gi1 complex with MP (2.5 Å) and LFT (3.2 Å) revealed that the two ligands engage distinct subpockets, and molecular dynamics simulations showed additional differences in the binding site that promote distinct active-state conformations on the intracellular side of the receptor where G proteins and β-arrestins bind. These observations highlight how drugs engaging different parts of the μOR orthosteric pocket can lead to distinct signaling outcomes. |
 リンク リンク |  Nat Chem Biol / Nat Chem Biol /  PubMed:36411392 / PubMed:36411392 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) |
| 解像度 | 2.5 - 3.2 Å |
| 構造データ | EMDB-25612, PDB-7t2g: EMDB-25613, PDB-7t2h: |
| 化合物 | 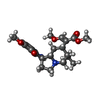 ChemComp-EIG:  ChemComp-CLR:  ChemComp-HOH: 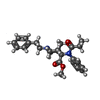 ChemComp-EID: |
| 由来 |
|
 キーワード キーワード |  MEMBRANE PROTEIN (膜タンパク質) / MEMBRANE PROTEIN (膜タンパク質) /  GPCR (Gタンパク質共役受容体) / GPCR (Gタンパク質共役受容体) /  mu-opioid receptor / lofentanil (LFT) / mitragynine pseudoindoxyl (MP) mu-opioid receptor / lofentanil (LFT) / mitragynine pseudoindoxyl (MP) |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について










