+検索条件
-Structure paper
| タイトル | A Structure-Based Model for the Complete Transcription Cycle of Influenza Polymerase. |
|---|---|
| ジャーナル・号・ページ | Cell, Vol. 181, Issue 4, Page 877-893.e21, Year 2020 |
| 掲載日 | 2020年5月14日 |
 著者 著者 | Joanna M Wandzik / Tomas Kouba / Manikandan Karuppasamy / Alexander Pflug / Petra Drncova / Jan Provaznik / Nayara Azevedo / Stephen Cusack /   |
| PubMed 要旨 | Influenza polymerase uses unique mechanisms to synthesize capped and polyadenylated mRNAs from the genomic viral RNA (vRNA) template, which is packaged inside ribonucleoprotein particles (vRNPs). ...Influenza polymerase uses unique mechanisms to synthesize capped and polyadenylated mRNAs from the genomic viral RNA (vRNA) template, which is packaged inside ribonucleoprotein particles (vRNPs). Here, we visualize by cryoelectron microscopy the conformational dynamics of the polymerase during the complete transcription cycle from pre-initiation to termination, focusing on the template trajectory. After exiting the active site cavity, the template 3' extremity rebinds into a specific site on the polymerase surface. Here, it remains sequestered during all subsequent transcription steps, forcing the template to loop out as it further translocates. At termination, the strained connection between the bound template 5' end and the active site results in polyadenylation by stuttering at uridine 17. Upon product dissociation, further conformational changes release the trapped template, allowing recycling back into the pre-initiation state. Influenza polymerase thus performs transcription while tightly binding to and protecting both template ends, allowing efficient production of multiple mRNAs from a single vRNP. |
 リンク リンク |  Cell / Cell /  PubMed:32304664 PubMed:32304664 |
| 手法 | EM (単粒子) / X線回折 |
| 解像度 | 2.41 - 3.52 Å |
| 構造データ | EMDB-10354, PDB-6szu: EMDB-10355, PDB-6szv: EMDB-10356, PDB-6t0n: EMDB-10357, PDB-6t0r: EMDB-10358, PDB-6t0s: EMDB-10359, PDB-6t0u: EMDB-10360, PDB-6t0v: EMDB-10361, PDB-6t0w: EMDB-10368, PDB-6t2c: EMDB-10603, PDB-6tw1:  PDB-6tu5: |
| 化合物 | 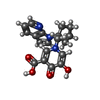 ChemComp-M4H:  ChemComp-MG:  ChemComp-POP:  ChemComp-HOH: 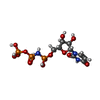 ChemComp-2KH: |
| 由来 |
|
 キーワード キーワード |  VIRAL PROTEIN (ウイルスタンパク質) / VIRAL PROTEIN (ウイルスタンパク質) /  Influenza (インフルエンザ) / Influenza (インフルエンザ) /  polymerase (ポリメラーゼ) / viral transcription / polymerase (ポリメラーゼ) / viral transcription /  RNA (リボ核酸) / RNA (リボ核酸) /  RNA-dependent RNA polymerase (RNA依存性RNAポリメラーゼ) RNA-dependent RNA polymerase (RNA依存性RNAポリメラーゼ) |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について
























