+検索条件
-Structure paper
| タイトル | Large-Scale Movements of IF3 and tRNA during Bacterial Translation Initiation. |
|---|---|
| ジャーナル・号・ページ | Cell, Vol. 167, Issue 1, Page 133-144.e13, Year 2016 |
| 掲載日 | 2016年9月22日 |
 著者 著者 | Tanweer Hussain / Jose L Llácer / Brian T Wimberly / Jeffrey S Kieft / V Ramakrishnan /   |
| PubMed 要旨 | In bacterial translational initiation, three initiation factors (IFs 1-3) enable the selection of initiator tRNA and the start codon in the P site of the 30S ribosomal subunit. Here, we report 11 ...In bacterial translational initiation, three initiation factors (IFs 1-3) enable the selection of initiator tRNA and the start codon in the P site of the 30S ribosomal subunit. Here, we report 11 single-particle cryo-electron microscopy (cryoEM) reconstructions of the complex of bacterial 30S subunit with initiator tRNA, mRNA, and IFs 1-3, representing different steps along the initiation pathway. IF1 provides key anchoring points for IF2 and IF3, thereby enhancing their activities. IF2 positions a domain in an extended conformation appropriate for capturing the formylmethionyl moiety charged on tRNA. IF3 and tRNA undergo large conformational changes to facilitate the accommodation of the formylmethionyl-tRNA (fMet-tRNA(fMet)) into the P site for start codon recognition. |
 リンク リンク |  Cell / Cell /  PubMed:27662086 / PubMed:27662086 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) |
| 解像度 | 3.55 - 9.7 Å |
| 構造データ | EMDB-4073, PDB-5lmn: EMDB-4074, PDB-5lmo: EMDB-4075, PDB-5lmp: EMDB-4076, PDB-5lmq: EMDB-4077, PDB-5lmr: EMDB-4078, PDB-5lms: EMDB-4079, PDB-5lmt: EMDB-4080: Structure of bacterial 30S-IF1-IF3-mRNA-tRNA translation pre-initiation complex, closed form (state-4)  EMDB-4081:  EMDB-4082: |
| 化合物 |  ChemComp-MG:  ChemComp-ZN: 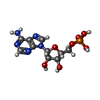 ChemComp-A: 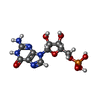 ChemComp-G: 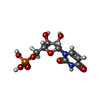 ChemComp-U: 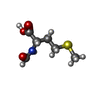 ChemComp-FME: |
| 由来 |
|
 キーワード キーワード |  RIBOSOME (リボソーム) / RIBOSOME (リボソーム) /  translation (翻訳 (生物学)) / translation (翻訳 (生物学)) /  initiation factors / 30S / IF1 / IF3 / PIC / initiation factors / 30S / IF1 / IF3 / PIC /  Thermus thermophilus (サーマス・サーモフィルス) / tRNAi / IF2 Thermus thermophilus (サーマス・サーモフィルス) / tRNAi / IF2 |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について























