+検索条件
-Structure paper
| タイトル | Structure and desensitization of AMPA receptor complexes with type II TARP γ5 and GSG1L. |
|---|---|
| ジャーナル・号・ページ | Mol Cell, Vol. 81, Issue 23, Page 4771-44783.e7, Year 2021 |
| 掲載日 | 2021年12月2日 |
 著者 著者 | Oleg Klykov / Shanti Pal Gangwar / Maria V Yelshanskaya / Laura Yen / Alexander I Sobolevsky /  |
| PubMed 要旨 | AMPA receptors (AMPARs) mediate the majority of excitatory neurotransmission. Their surface expression, trafficking, gating, and pharmacology are regulated by auxiliary subunits. Of the two types of ...AMPA receptors (AMPARs) mediate the majority of excitatory neurotransmission. Their surface expression, trafficking, gating, and pharmacology are regulated by auxiliary subunits. Of the two types of TARP auxiliary subunits, type I TARPs assume activating roles, while type II TARPs serve suppressive functions. We present cryo-EM structures of GluA2 AMPAR in complex with type II TARP γ5, which reduces steady-state currents, increases single-channel conductance, and slows recovery from desensitization. Regulation of AMPAR function depends on its ligand-binding domain (LBD) interaction with the γ5 head domain. GluA2-γ5 complex shows maximum stoichiometry of two TARPs per AMPAR tetramer, being different from type I TARPs but reminiscent of the auxiliary subunit GSG1L. Desensitization of both GluA2-GSG1L and GluA2-γ5 complexes is accompanied by rupture of LBD dimer interface, while GluA2-γ5 but not GluA2-GSG1L LBD dimers remain two-fold symmetric. Different structural architectures and desensitization mechanisms of complexes with auxiliary subunits endow AMPARs with broad functional capabilities. |
 リンク リンク |  Mol Cell / Mol Cell /  PubMed:34678168 / PubMed:34678168 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) |
| 解像度 | 3.3 - 4.4 Å |
| 構造データ | EMDB-24748, PDB-7ryy: EMDB-24749, PDB-7ryz: EMDB-24750, PDB-7rz4: EMDB-24751, PDB-7rz5: EMDB-24752, PDB-7rz6: EMDB-24753, PDB-7rz7: EMDB-24754, PDB-7rz8: EMDB-24755, PDB-7rz9: EMDB-24756, PDB-7rza: |
| 化合物 |  ChemComp-PCW:  ChemComp-GLU: 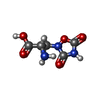 ChemComp-QUS: 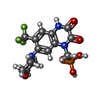 ChemComp-ZK1:  ChemComp-NAG: |
| 由来 |
|
 キーワード キーワード |  MEMBRANE PROTEIN (膜タンパク質) / MEMBRANE PROTEIN (膜タンパク質) /  AMPA receptor (AMPA型グルタミン酸受容体) / AMPA receptor (AMPA型グルタミン酸受容体) /  ion channel (イオンチャネル) / ion channel (イオンチャネル) /  neurotransmission / neurotransmission /  synapse (シナプス) / TARP gamma-5 / GSG1L synapse (シナプス) / TARP gamma-5 / GSG1L |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について






















