+検索条件
-Structure paper
| タイトル | Structural Basis of Functional Transitions in Mammalian NMDA Receptors. |
|---|---|
| ジャーナル・号・ページ | Cell, Vol. 182, Issue 2, Page 357-371.e13, Year 2020 |
| 掲載日 | 2020年7月23日 |
 著者 著者 | Tsung-Han Chou / Nami Tajima / Annabel Romero-Hernandez / Hiro Furukawa /  |
| PubMed 要旨 | Excitatory neurotransmission meditated by glutamate receptors including N-methyl-D-aspartate receptors (NMDARs) is pivotal to brain development and function. NMDARs are heterotetramers composed of ...Excitatory neurotransmission meditated by glutamate receptors including N-methyl-D-aspartate receptors (NMDARs) is pivotal to brain development and function. NMDARs are heterotetramers composed of GluN1 and GluN2 subunits, which bind glycine and glutamate, respectively, to activate their ion channels. Despite importance in brain physiology, the precise mechanisms by which activation and inhibition occur via subunit-specific binding of agonists and antagonists remain largely unknown. Here, we show the detailed patterns of conformational changes and inter-subunit and -domain reorientation leading to agonist-gating and subunit-dependent competitive inhibition by providing multiple structures in distinct ligand states at 4 Å or better. The structures reveal that activation and competitive inhibition by both GluN1 and GluN2 antagonists occur by controlling the tension of the linker between the ligand-binding domain and the transmembrane ion channel of the GluN2 subunit. Our results provide detailed mechanistic insights into NMDAR pharmacology, activation, and inhibition, which are fundamental to the brain physiology. |
 リンク リンク |  Cell / Cell /  PubMed:32610085 / PubMed:32610085 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) / X線回折 |
| 解像度 | 2.092 - 4.39 Å |
| 構造データ | EMDB-21673, PDB-6whr: EMDB-21674, PDB-6whs: EMDB-21675, PDB-6wht: EMDB-21676, PDB-6whu: EMDB-21677, PDB-6whv: EMDB-21678, PDB-6whw: EMDB-21679, PDB-6whx: EMDB-21680, PDB-6why: EMDB-21681, PDB-6wi0: EMDB-21682, PDB-6wi1:  PDB-6usu:  PDB-6usv: |
| 化合物 | 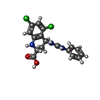 ChemComp-QGM:  ChemComp-GLU:  ChemComp-HOH:  ChemComp-GLY: 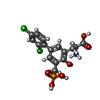 ChemComp-QGP:  ChemComp-GOL:  ChemComp-NAG: |
| 由来 |
|
 キーワード キーワード | METAL TRANSPORT / NMDARs / LBD /  Ion channels (イオンチャネル) / Ion channels (イオンチャネル) /  MEMBRANE PROTEIN (膜タンパク質) / MEMBRANE PROTEIN (膜タンパク質) /  Ligand-gated ion channels (リガンド依存性イオンチャネル) / Ligand-gated ion channels (リガンド依存性イオンチャネル) /  Ionotropic glutamate receptor / GluN1 antagonist / GluN2B antagonist Ionotropic glutamate receptor / GluN1 antagonist / GluN2B antagonist |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について
























