-検索条件
-検索結果
検索 (著者・登録者: suzuki & s)の結果344件中、1から50件目までを表示しています

EMDB-36933: 
Cryo-EM map of SV2A in complex with brivaracetam and BoNT/A2 Hc

EMDB-36934: 
Local map of SV2A LD4-BoNT/A2 Hc from SV2A-BoNT/A2 Hc-brivaracetam complex

EMDB-36392: 
Cryo-EM structure of SV2A in complex with BoNT/A2 Hc and levetiracetam

EMDB-36394: 
Cryo-EM structure of SV2A LD4 in complex with BoNT/A2 Hc in the SV2A-levetiracetam-BoNT/A2 Hc complex

EMDB-36395: 
Cryo-EM structure of SV2A in complex with BoNT/A2 Hc

EMDB-36396: 
Cryo-EM structure of SV2A in complex with BoNT/A2 Hc

EMDB-36397: 
Cryo-EM structure of SV2A dimer in complex with BoNT/A2 Hc and levetiracetam

EMDB-36398: 
Cryo-EM structure of SV2A dimer in complex levetiracetam

EMDB-36616: 
Cryo-EM structure of SV2A in complex with BoNT/A2 Hc and levetiracetam

EMDB-36617: 
Cryo-EM structure of SV2A in complex with BoNT/A2 Hc

EMDB-36935: 
Cryo-EM structure of SV2A in complex with BoNT/A2 Hc and brivaracetam

PDB-8jlc: 
Cryo-EM structure of SV2A in complex with BoNT/A2 Hc and levetiracetam

PDB-8jle: 
Cryo-EM structure of SV2A LD4 in complex with BoNT/A2 Hc in the SV2A-levetiracetam-BoNT/A2 Hc complex

PDB-8jlf: 
Cryo-EM structure of SV2A in complex with BoNT/A2 Hc

PDB-8jlg: 
Cryo-EM structure of SV2A in complex with BoNT/A2 Hc

PDB-8jlh: 
Cryo-EM structure of SV2A dimer in complex with BoNT/A2 Hc and levetiracetam

PDB-8jli: 
Cryo-EM structure of SV2A dimer in complex levetiracetam

PDB-8js8: 
Cryo-EM structure of SV2A in complex with BoNT/A2 Hc and levetiracetam

PDB-8js9: 
Cryo-EM structure of SV2A in complex with BoNT/A2 Hc

PDB-8k77: 
Cryo-EM structure of SV2A in complex with BoNT/A2 Hc and brivaracetam

EMDB-38986: 
P-glycoprotein in complex with UIC2 Fab and triple elacridar molecules in LMNG detergent

EMDB-38987: 
P-glycoprotein in complex with UIC2 Fab and triple elacridar molecules in nanodisc

PDB-8y6h: 
P-glycoprotein in complex with UIC2 Fab and triple elacridar molecules in LMNG detergent

PDB-8y6i: 
P-glycoprotein in complex with UIC2 Fab and triple elacridar molecules in nanodisc

EMDB-35001: 
E. coli 70S ribosome complexed with tRNA_Ile2 bearing L34 and t6A37 in classical state

EMDB-35020: 
E. coli 70S ribosome complexed with H. marismortui tRNA_Ile2 bearing agm2C34 in classical state

EMDB-35022: 
E. coli 70S ribosome complexed with tRNA_Ile2 bearing L34 and ct6A37 in classical state

PDB-8hsp: 
E. coli 70S ribosome complexed with tRNA_Ile2 bearing L34 and t6A37 in classical state

PDB-8htz: 
E. coli 70S ribosome complexed with H. marismortui tRNA_Ile2 bearing agm2C34 in classical state

PDB-8hu1: 
E. coli 70S ribosome complexed with tRNA_Ile2 bearing L34 and ct6A37 in classical state

EMDB-37480: 
PSI-LHCI of the red alga Cyanidium caldarium RK-1 (NIES-2137)

PDB-8wey: 
PSI-LHCI of the red alga Cyanidium caldarium RK-1 (NIES-2137)
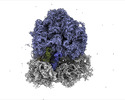
EMDB-35411: 
Dibekacin-bound E.coli 70S ribosome in the PURE system
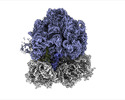
EMDB-35412: 
Arbekacin-bound E.coli 70S ribosome in the PURE system
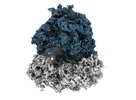
EMDB-35413: 
Dibekacin-added human 80S ribosome
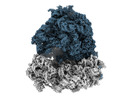
EMDB-35414: 
Arbekacin-added human 80S ribosome

PDB-8ifb: 
Dibekacin-bound E.coli 70S ribosome in the PURE system

PDB-8ifc: 
Arbekacin-bound E.coli 70S ribosome in the PURE system

PDB-8ifd: 
Dibekacin-added human 80S ribosome

PDB-8ife: 
Arbekacin-added human 80S ribosome

EMDB-41837: 
The structure of the PP2A-B56Delta holoenzyme mutant - E197K

PDB-8u1x: 
The structure of the PP2A-B56Delta holoenzyme mutant - E197K

EMDB-42018: 
The structure of the PP2A-B56Delta holoenzyme mutant - E197K

PDB-8u89: 
The structure of the PP2A-B56Delta holoenzyme mutant - E197K

EMDB-36724: 
Structure of the SARS-CoV-2 XBB.1.5 spike glycoprotein (closed state 1)

EMDB-36726: 
Structure of the SARS-CoV-2 XBB.1.5 spike glycoprotein (closed state 2)

EMDB-36727: 
Structure of SARS-CoV-2 XBB.1.5 spike glycoprotein in complex with ACE2 (1-up state)

EMDB-36728: 
Structure of SARS-CoV-2 XBB.1.5 spike glycoprotein in complex with ACE2 (2-up state)

EMDB-36729: 
Structure of SARS-CoV-2 XBB.1.5 spike RBD in complex with ACE2

EMDB-35029: 
SARS-CoV2 spike protein with ACE2, no ACE2 binding.
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
