-検索条件
-検索結果
検索 (著者・登録者: sebastian & e)の結果983件中、1から50件目までを表示しています

EMDB-17213: 
Human Mitochondrial Lon Y186F Mutant ADP Bound

EMDB-17214: 
Human Mitochondrial Lon Y186E Mutant ADP Bound

PDB-8ovf: 
Human Mitochondrial Lon Y186F Mutant ADP Bound

PDB-8ovg: 
Human Mitochondrial Lon Y186E Mutant ADP Bound

EMDB-18214: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex - hexameric assembly
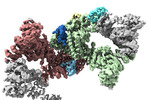
EMDB-18216: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex in unneddylated and neddylated conformation - focused cullin dimer
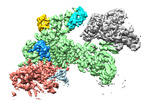
EMDB-18217: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex in unneddylated and neddylated conformation - focused on E2-like density

EMDB-18218: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex in unneddylated and neddylated conformation - focused dimeric core
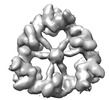
EMDB-18220: 
Structure of the hexameric CUL9-RBX1 complex with deletion of CUL9 CPH domain
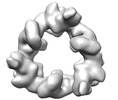
EMDB-18221: 
Structure of the hexameric CUL9-RBX1 complex with deletion of CUL9 DOC domain
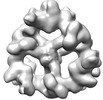
EMDB-18222: 
Structure of the hexameric CUL9-RBX1 complex with deletion of CUL9 ARM9 domain
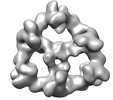
EMDB-18223: 
Structure of the hexameric CUL9-RBX1 complex with deletion of CUL9 ARIH-RBR element

EMDB-19179: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex in unneddylated conformation - symmetry expanded unneddylated dimer

PDB-8q7e: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex - hexameric assembly

PDB-8q7h: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex in unneddylated and neddylated conformation - focused cullin dimer

PDB-8rhz: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex in unneddylated conformation - symmetry expanded unneddylated dimer

EMDB-16923: 
Human Mitochondrial Lon Y394F Mutant ADP Bound

EMDB-16970: 
Human Mitochondrial Lon Y186E Mutant ADP Bound

PDB-8oka: 
Human Mitochondrial Lon Y394F Mutant ADP Bound

PDB-8om7: 
Human Mitochondrial Lon Y186E Mutant ADP Bound

EMDB-16915: 
Human Mitochondrial Lon Y394E Mutant ADP Bound

PDB-8ojl: 
Human Mitochondrial Lon Y394E Mutant ADP Bound

EMDB-17509: 
Cryo-EM structure of CAK in complex with inhibitor BS-181

EMDB-17510: 
Cryo-EM structure of CAK in complex with inhibitor BS-194

EMDB-17512: 
Cryo-EM structure of CAK in complex with inhibitor ICEC0510-R

EMDB-17513: 
Cryo-EM structure of CAK in complex with inhibitor ICEC0510-S

EMDB-17514: 
Cryo-EM structure of CAK in complex with inhibitor ICEC0574

EMDB-17515: 
Cryo-EM structure of CAK in complex with inhibitor ICEC0768

EMDB-17516: 
Cryo-EM structure of CAK in complex with inhibitor ICEC0829

EMDB-17517: 
Cryo-EM structure of CAK in complex with inhibitor ICEC0880 (ring-up conformation)

EMDB-17518: 
Cryo-EM structure of CAK in complex with inhibitor ICEC0880 (ring-down conformation)

EMDB-17519: 
Cryo-EM structure of CAK in complex with inhibitor ICEC0914

EMDB-17520: 
Cryo-EM structure of CAK in complex with inhibitor ICEC0943

EMDB-17521: 
Cryo-EM structure of CAK in complex with inhibitor dinaciclib

EMDB-17522: 
Cryo-EM structure of CAK with averaged inhibitor density

EMDB-17523: 
Cryo-EM structure of apo CAK

EMDB-17524: 
Cryo-EM map of inhibitor-bound CAK (Krios G4 performance comparison dataset)

EMDB-17525: 
Cryo-EM map of inhibitor-bound CAK (Glacios G2 performance comparison dataset)

EMDB-17526: 
Cryo-EM map of inhibitor-bound CAK (+EF performance comparison dataset)

EMDB-17527: 
Cryo-EM map of inhibitor-bound CAK (-EF performance comparison dataset)

EMDB-17536: 
Cryo-EM structure of CDK7 subunit of CAK in complex with inhibitor LDC4297

EMDB-17754: 
Cryo-EM structure of CAK in complex with inhibitor CT7030

PDB-8p6w: 
Cryo-EM structure of CAK in complex with inhibitor BS-181

PDB-8p6x: 
Cryo-EM structure of CAK in complex with inhibitor BS-194

PDB-8p6z: 
Cryo-EM structure of CAK in complex with inhibitor ICEC0510-R

PDB-8p70: 
Cryo-EM structure of CAK in complex with inhibitor ICEC0510-S

PDB-8p71: 
Cryo-EM structure of CAK in complex with inhibitor ICEC0574

PDB-8p72: 
Cryo-EM structure of CAK in complex with inhibitor ICEC0768

PDB-8p73: 
Cryo-EM structure of CAK in complex with inhibitor ICEC0829

PDB-8p74: 
Cryo-EM structure of CAK in complex with inhibitor ICEC0880 (ring-up conformation)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
