-検索条件
-検索結果
検索 (著者・登録者: sanchez & r)の結果262件中、1から50件目までを表示しています

EMDB-16103: 
Human serotonin 5-HT3A receptor (apo, active/distorted conformation)

EMDB-16104: 
Human serotonin 5-HT3A receptor in complex with vortioxetine (detergent, ECD only, active/distorted conformation)

EMDB-16105: 
Human serotonin 5-HT3A receptor in complex with vortioxetine (nanodiscs, ECD, active/distorted conformation)

PDB-8bl8: 
Human serotonin 5-HT3A receptor (apo, active/distorted conformation)

PDB-8bla: 
Human serotonin 5-HT3A receptor in complex with vortioxetine (detergent, ECD only, active/distorted conformation)

PDB-8blb: 
Human serotonin 5-HT3A receptor in complex with vortioxetine (nanodiscs, ECD, active/distorted conformation)
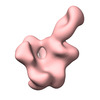
EMDB-42464: 
chEnv TTT protein in complex with 43A2 Fab

EMDB-42468: 
chEnv TTT protein in complex with CM01A Fab
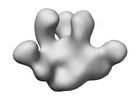
EMDB-43664: 
Triple tandem trimer immunogens for HIV-1 and influenza nucleic acid-based vaccines
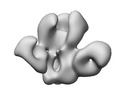
EMDB-43665: 
Triple tandem trimer immunogens for HIV-1 and influenza nucleic acid-based vaccines (cH125 TTT)

EMDB-43666: 
Triple tandem trimer immunogens for HIV-1 and influenza nucleic acid-based vaccines (H2/1 GCN4)

EMDB-43668: 
Triple tandem trimer immunogens for HIV-1 and influenza nucleic acid-based vaccines (H5/1 GCN4)

EMDB-43669: 
Triple tandem trimer immunogens for HIV-1 and influenza nucleic acid-based vaccines. H5 GCN4

EMDB-16916: 
Bipartite interaction of TOPBP1 with the GINS complex

PDB-8ok2: 
Bipartite interaction of TOPBP1 with the GINS complex

EMDB-15689: 
Mouse serotonin 5-HT3A receptor in complex with vortioxetine
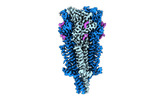
EMDB-15699: 
Human serotonin 5-HT3A receptor (apo, resting conformation)

PDB-8aw2: 
Mouse serotonin 5-HT3A receptor in complex with vortioxetine

PDB-8axd: 
Human serotonin 5-HT3A receptor (apo, resting conformation)

EMDB-19250: 
Pseudoatomic model of a second-order Sierpinski triangle formed by the citrate synthase from Synechococcus elongatus

EMDB-19251: 
Structure of a first order Sierpinski triangle formed by the H369R mutant of the citrate synthase from Synechococcus elongatus

EMDB-15474: 
In situ cryo-electron tomogram of an intact primary neuronal process

EMDB-15454: 
Subtomogram average of bound microtubule inner protein 2

EMDB-15457: 
Subtomogram average of floating microtubule inner protein 5

EMDB-15463: 
Subtomogram average of floating microtubule inner protein 12

EMDB-15456: 
In situ subtomogram average of bound microtubule inner protein 4 (bMIP4)

EMDB-15459: 
In situ subtomogram average of floating microtubule inner protein 7

EMDB-15464: 
In situ subtomogram average of floating microtubule inner protein 13 (fMIP13)

EMDB-15458: 
Subtomogram average of floating microtubule inner protein 6

EMDB-15461: 
Subtomogram average of floating microtubule inner protein 10

EMDB-15472: 
In situ subtomogram average of floating microtubule inner protein 8

EMDB-15469: 
Subtomogram average of murine 80S ribosome

EMDB-15460: 
Subtomogram average of floating microtubule inner protein 9

EMDB-15455: 
Subtomogram average of bound microtubule inner protein 3

EMDB-15444: 
Subtomogram average of floating microtubule inner protein 4

EMDB-15467: 
In situ subtomogram average of floating microtubule inner protein 15 (fMIP15)

EMDB-15470: 
In situ subtomogram average of primary neuronal microtubules

EMDB-15466: 
In situ subtomogram average of floating microtubule inner protein 14 (fMIP14)

EMDB-15453: 
In situ subtomogram average of bound microtubule inner protein 1 (bMIP1)

EMDB-15468: 
In situ subtomogram average of floating microtubule inner protein 16 (fMIP16)

EMDB-15462: 
Subtomogram average of floating microtubule inner protein 11

EMDB-15440: 
In situ cryo-electron tomogram of an intact murine P19 cellular process.

EMDB-15437: 
In situ cryo-electron tomogram of an intact human induced pluripotent stem cell derived neuronal process

EMDB-15438: 
In situ cryo-electron tomogram of an intact differentiated P19 neuronal process
EMDB-15442: 
In situ cryo-electron tomogram of an intact pluripotent P19 cellular process after nocodazole treatment and washout.

EMDB-15443: 
In situ cryo-electron tomogram of a Taxol treated intact pluripotent P19 cell.

EMDB-15439: 
Subtomogram average of floating microtubule inner protein 2 (fMIP2)

EMDB-15436: 
In situ subtomogram average of floating microtubule inner protein 3 (fMIP3)

EMDB-15441: 
In situ subtomogram average of floating microtubule inner protein 1 (fMIP1)
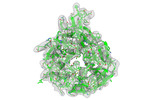
EMDB-15411: 
Single particle structure of Atg18-WT
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
