-検索条件
-検索結果
検索 (著者・登録者: noel & ea)の結果51件中、1から50件目までを表示しています

EMDB-16493: 
cryo-EM structure of BG505 SOSIP.664 HIV-1 Env trimer in complex with bNAbs EPTC112 and 3BNC117

EMDB-35326: 
Cryo-EM reconstruction of PBCV-1 capsid block 1

EMDB-35490: 
Cryo-EM reconstruction of PBCV-1 capsid block 2

EMDB-35585: 
Cryo-EM reconstruction of PBCV-1 capsid block 3

EMDB-35586: 
Cryo-EM reconstruction of PBCV-1 capsid block 4

EMDB-35589: 
Cryo-EM reconstruction of PBCV-1 capsid block 5

EMDB-35593: 
Cryo-EM reconstruction of PBCV-1 capsid block 6

EMDB-35594: 
Cryo-EM reconstruction of PBCV-1 capsid block 8

EMDB-35644: 
Cryo-EM reconstruction of PBCV-1 capsid block 9

EMDB-35645: 
Cryo-EM reconstruction of PBCV-1 capsid block 7

EMDB-35646: 
Cryo-EM reconstruction of PBCV-1 capsid block 10

EMDB-35647: 
Cryo-EM reconstruction of PBCV-1 capsid block 11

EMDB-35648: 
Cryo-EM reconstruction of PBCV-1 capsid block 12

EMDB-35650: 
Cryo-EM reconstruction of PBCV-1 capsid block 13

EMDB-35656: 
Cryo-EM reconstruction of PBCV-1 capsid block 14

EMDB-35659: 
Cryo-EM reconstruction of PBCV-1 capsid block 15

EMDB-35695: 
Cryo-EM reconstruction of PBCV-1 capsid block 16

EMDB-35954: 
Cryo-EM reconstruction of PBCV-1 capsid block 17

EMDB-35955: 
Cryo-EM reconstruction of PBCV-1 capsid block 18

EMDB-35956: 
Cryo-EM reconstruction of PBCV-1 capsid block 19

EMDB-35957: 
Cryo-EM reconstruction of PBCV-1 capsid block 20

EMDB-35958: 
Cryo-EM reconstruction of PBCV-1 capsid block 21

EMDB-35959: 
Cryo-EM reconstruction of PBCV-1 capsid block 22

EMDB-35960: 
Cryo-EM reconstruction of PBCV-1 capsid block 23
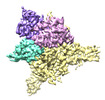
EMDB-27898: 
Cryo-EM structure of human glycerol-3-phosphate acyltransferase 1 (GPAT1) in complex with 2-oxohexadecyl-CoA
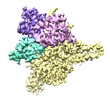
EMDB-27899: 
Cryo-EM structure of human glycerol-3-phosphate acyltransferase 1 (GPAT1) in complex with CoA and palmitoyl-LPA

PDB-8e4y: 
Cryo-EM structure of human glycerol-3-phosphate acyltransferase 1 (GPAT1) in complex with 2-oxohexadecyl-CoA

PDB-8e50: 
Cryo-EM structure of human glycerol-3-phosphate acyltransferase 1 (GPAT1) in complex with CoA and palmitoyl-LPA

EMDB-34438: 
Near-atomic structure of five-fold averaged PBCV-1 capsid

EMDB-34439: 
Five-fold averaged cryo-EM reconstruction of PBCV-1 before applying the "block-based" reconstruction method

EMDB-27203: 
IMM20190 Fab complex with SARS-CoV-2 Spike Trimer

EMDB-27192: 
IMM20253 Fab complex with Trimer Spike protein of SARS-CoV-2 virus
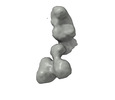
EMDB-27193: 
IMM20253 Fab complex with Spike monomer
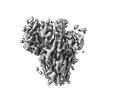
EMDB-27204: 
IMM20184 Fab complex with SARS-CoV-2 Spike Trimer

EMDB-27081: 
IMM20184 and IMM20253 Fabs in ternary complex with SARS-CoV-2 receptor binding domain

EMDB-23118: 
Orexin Receptor 2 (OX2R) in Complex with G Protein and Natural Peptide-Agonist Orexin B (OxB)

EMDB-23119: 
Orexin Receptor 2 (OX2R) in Complex with G Protein and Small-Molecule Agonist Compound 1

PDB-7l1u: 
Orexin Receptor 2 (OX2R) in Complex with G Protein and Natural Peptide-Agonist Orexin B (OxB)

PDB-7l1v: 
Orexin Receptor 2 (OX2R) in Complex with G Protein and Small-Molecule Agonist Compound 1

EMDB-4584: 
Structure and assembly of the mitochondrial membrane remodelling GTPase Mgm1
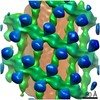
EMDB-10062: 
Structure of s-Mgm1 decorating the outer surface of tubulated lipid membranes
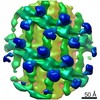
EMDB-10063: 
Structure of s-Mgm1 decorating the outer surface of tubulated lipid membranes in the GTPgammaS bound state

EMDB-10064: 
Structure of s-Mgm1 decorating the inner surface of tubulated lipid membranes

EMDB-10065: 
Structure of s-Mgm1 decorating the inner surface of tubulated lipid membranes in the GTPgammaS bound state

PDB-6rzt: 
Structure of s-Mgm1 decorating the outer surface of tubulated lipid membranes

PDB-6rzu: 
Structure of s-Mgm1 decorating the outer surface of tubulated lipid membranes in the GTPgammaS bound state

PDB-6rzv: 
Structure of s-Mgm1 decorating the inner surface of tubulated lipid membranes

PDB-6rzw: 
Structure of s-Mgm1 decorating the inner surface of tubulated lipid membranes in the GTPgammaS bound state

EMDB-3896: 
Cryo-EM Structure of Saccharomyces cerevisiae Target of Rapamycin Complex 2

PDB-6emk: 
Cryo-EM Structure of Saccharomyces cerevisiae Target of Rapamycin Complex 2
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
