-検索条件
-検索結果
検索 (著者・登録者: li & fan & chen)の結果1,982件中、1から50件目までを表示しています

EMDB-37342: 
Structural mechanism of inhibition of the Rho transcription termination factor by Rof

PDB-8w8d: 
Structural mechanism of inhibition of the Rho transcription termination factor by Rof

EMDB-36849: 
Nipah virus Attachment glycoprotein with 41-6 antibody fragment

PDB-8k3c: 
Nipah virus Attachment glycoprotein with 41-6 antibody fragment

EMDB-36760: 
Cryo-EM structure of conformation 1 of complex of Nipah virus attachment glycoprotein G with 1E5 neutralizing antibody

EMDB-36761: 
Cryo-EM structure of conformation 2 of complex of Nipah virus attachment G with 1E5 neutralizing antibody

EMDB-43991: 
Cryo-EM structure of apo state human Cav3.2

EMDB-43992: 
Cryo-EM structure of human Cav3.2 with TTA-A2

EMDB-43993: 
Cryo-EM structure of human Cav3.2 with TTA-P2

EMDB-43994: 
Cryo-EM structure of human Cav3.2 with ML218

EMDB-43995: 
Cryo-EM structure of human Cav3.2 with ACT-709478

PDB-9ayg: 
Cryo-EM structure of apo state human Cav3.2

PDB-9ayh: 
Cryo-EM structure of human Cav3.2 with TTA-A2

PDB-9ayj: 
Cryo-EM structure of human Cav3.2 with TTA-P2

PDB-9ayk: 
Cryo-EM structure of human Cav3.2 with ML218

PDB-9ayl: 
Cryo-EM structure of human Cav3.2 with ACT-709478

EMDB-43931: 
CryoEM structure of activated CRAF/MEK/14-3-3 complex with NST-628

EMDB-43932: 
Activated CRAF/MEK heterotetramer from focused refinement of CRAF/MEK/14-3-3 complex

PDB-9axa: 
CryoEM structure of activated CRAF/MEK/14-3-3 complex with NST-628

PDB-9axc: 
Activated CRAF/MEK heterotetramer from focused refinement of CRAF/MEK/14-3-3 complex
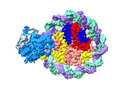
EMDB-37529: 
Structure of DDM1-nucleosome complex in the apo state

EMDB-37533: 
Structure of DDM1-nucleosome complex in ADP state
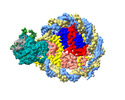
EMDB-37535: 
Structure of DDM1-nucleosome complex in ADP-BeFx state

EMDB-37537: 
Structure of DDM1-nucleosome complex in the ADP-BeFx state with DDM1 bound to SHL2 and SHL-2
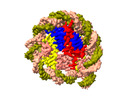
EMDB-37538: 
Structure of nucleosome core particle of Arabidopsis thaliana

PDB-8wh5: 
Structure of DDM1-nucleosome complex in the apo state

PDB-8wh8: 
Structure of DDM1-nucleosome complex in ADP state

PDB-8wh9: 
Structure of DDM1-nucleosome complex in ADP-BeFx state

PDB-8wha: 
Structure of DDM1-nucleosome complex in the ADP-BeFx state with DDM1 bound to SHL2 and SHL-2

PDB-8whb: 
Structure of nucleosome core particle of Arabidopsis thaliana

EMDB-38200: 
Cryo-EM structure of OSCA1.2-liposome-inside-in open state

EMDB-38503: 
Cryo-EM structure of OSCA1.2-liposome-inside-out closed state

EMDB-38611: 
Cryo-EM structure of OSCA3.1-1.1ver(Y367N-G454S-Y458I)-open/open state

EMDB-38612: 
Cryo-EM structure of OSCA3.1-1.1ver(Y367N-G454S-Y458I)-open/'desensitized' state

EMDB-38614: 
Cryo-EM structure of OSCA1.2-DOPC-1:20-contracted1 state

EMDB-38615: 
Cryo-EM structure of OSCA1.2-DOPC-1:20-contracted2 state

EMDB-38721: 
Cryo-EM structure of OSCA1.2-DOPC-1:20-expanded state

EMDB-38722: 
Cryo-EM structure of OSCA1.2-DOPC-1:50-betaCD state

EMDB-38723: 
Cryo-EM structure of OSCA3.1-2E(R611E-R619E)-closed/open state

EMDB-38724: 
Cryo-EM structure of OSCA3.1-2E(R611E-R619E)-closed/'desensitized' state

EMDB-38725: 
Cryo-EM structure of OSCA3.1-GDN state

EMDB-38726: 
Cryo-EM structure of OSCA3.1-liposome-inside-in state

EMDB-38727: 
Cryo-EM structure of OSCA1.2-V335W-DDM state

EMDB-38728: 
Cryo-EM structure of OSCA1.2-DOPC-1:50-contracted state

EMDB-38729: 
Cryo-EM structure of OSCA1.2-DOPC-1:50-expanded state

EMDB-38730: 
Cryo-EM structure of TMEM63B-Digitonin state

PDB-8xaj: 
Cryo-EM structure of OSCA1.2-liposome-inside-in open state

PDB-8xng: 
Cryo-EM structure of OSCA1.2-liposome-inside-out closed state

PDB-8xry: 
Cryo-EM structure of OSCA3.1-1.1ver(Y367N-G454S-Y458I)-open/open state

PDB-8xs0: 
Cryo-EM structure of OSCA3.1-1.1ver(Y367N-G454S-Y458I)-open/'desensitized' state
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
