-検索条件
-検索結果
検索 (著者・登録者: kaji & a)の結果56件中、1から50件目までを表示しています

EMDB-37480: 
PSI-LHCI of the red alga Cyanidium caldarium RK-1 (NIES-2137)

PDB-8wey: 
PSI-LHCI of the red alga Cyanidium caldarium RK-1 (NIES-2137)

EMDB-34981: 
Cryo-EM structure of human NTCP-myr-preS1-YN9048Fab complex

EMDB-34982: 
Cryo-EM structure of human NTCP-myr-preS1-YN9016Fab complex

PDB-8hrx: 
Cryo-EM structure of human NTCP-myr-preS1-YN9048Fab complex

PDB-8hry: 
Cryo-EM structure of human NTCP-myr-preS1-YN9016Fab complex
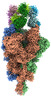
EMDB-33972: 
Cryo-EM structure of the SARS-CoV-2 spike protein (3-up RBD) bound to neutralizing antibody CSW1-1805
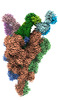
EMDB-33973: 
Cryo-EM structure of the SARS-CoV-2 spike protein (1-up RBD) bound to neutralizing antibody CSW2-1353
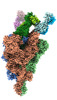
EMDB-33974: 
Cryo-EM structure of the SARS-CoV-2 spike protein (2-up RBD) bound to neutralizing antibody CSW2-1353
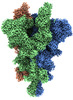
EMDB-33975: 
Cryo-EM structure of the SARS-CoV-2 spike protein (2-up RBD) with C480A mutation

EMDB-33593: 
Structure of the Anabaena PSI-monomer-IsiA supercomplex

PDB-7y3f: 
Structure of the Anabaena PSI-monomer-IsiA supercomplex

EMDB-31526: 
human NTCP in complex with YN69083 Fab

PDB-7fci: 
human NTCP in complex with YN69083 Fab

EMDB-31455: 
Cryo-EM structure of a primordial cyanobacterial photosystem I

EMDB-31062: 
Structure of monomeric photosystem II

PDB-7eda: 
Structure of monomeric photosystem II

EMDB-30547: 
Cryo-EM Structure of PSII at 1.95 angstrom resolution

EMDB-30548: 
Cryo-EM Structure of PSII at 2.08 angstrom resolution

EMDB-30549: 
Cryo-EM Structure of PSII at 2.22 angstrom resolution

EMDB-30550: 
Cryo-EM Structure of PSII at 2.20 angstrom resolution

PDB-7d1t: 
Cryo-EM Structure of PSII at 1.95 angstrom resolution

PDB-7d1u: 
Cryo-EM Structure of PSII at 2.08 angstrom resolution

EMDB-12160: 
Cilia from MOT7 deletion mutant of Chlamydomonas

EMDB-12161: 
Chlamydomonas cilia with MOT7-BCCP labeled

EMDB-12162: 
Microtubule doublet structure from WT Chlamydomonas as a control for MOT7 mutants

EMDB-9908: 
Structure of PSI-isiA supercomplex from Thermosynechococcus vulcanus

PDB-6k33: 
Structure of PSI-isiA supercomplex from Thermosynechococcus vulcanus

EMDB-10262: 
Yeast 80S ribosome stalled on SDD1 mRNA.

EMDB-10315: 
The cryo-EM structure of SDD1-stalled collided trisome.

PDB-6snt: 
Yeast 80S ribosome stalled on SDD1 mRNA.

PDB-6sv4: 
The cryo-EM structure of SDD1-stalled collided trisome.

EMDB-9530: 
Cryo-EM structure of the model post-termination complex (PoTC) in the rotated state

EMDB-9531: 
Cryo-EM structure of the model post-termination complex (PoTC) in the unrotated state

EMDB-9532: 
Cryo-EM structure of the model post-termination complex (PoTC) after the ribosome recycling reaction

EMDB-9533: 
Cryo-EM structure of the 70S ribosome from polysome

EMDB-8096: 
The Architecture of the Cytoplasmic Region of Type III Secretion Systems

EMDB-8121: 
The Architecture of the Cytoplasmic Region of Type III Secretion Systems

EMDB-8122: 
The Architecture of the Cytoplasmic Region of Type III Secretion Systems

EMDB-1915: 
Initial binding position of RRF on the post-termination complex

EMDB-1916: 
Initial binding conformation of RRF on the post-termination complex

EMDB-1917: 
Intermediate binding positions of RRF and EF-G on the post-termination complex

EMDB-1918: 
Binding conformations and positions of RRF and EF-G during intermediate state of ribosome recycling

PDB-3j0d: 
Models for the T. thermophilus ribosome recycling factor bound to the E. coli post-termination complex

PDB-3j0e: 
Models for the T. thermophilus ribosome recycling factor and the E. coli elongation factor G bound to the E. coli post-termination complex

EMDB-5254: 
WT Dam1 complex assembled into a ring around a microtubule

PDB-3iyo: 
Cryo-EM model of virion-sized HEV virion-sized capsid

EMDB-5173: 
Cryo-EM structure of virion-sized hepatitis E virus-like particle

EMDB-1369: 
Progression of the ribosome recycling factor through the ribosome dissociates the two ribosomal subunits.

EMDB-1370: 
Progression of the ribosome recycling factor through the ribosome dissociates the two ribosomal subunits.
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
