-検索条件
-検索結果
検索 (著者・登録者: jeffrey & c)の結果878件中、1から50件目までを表示しています
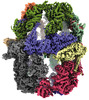
EMDB-19477: 
Saccharomyces cerevisiae FAS type I

EMDB-19489: 
Tobacco mosaic virus from scanning transmission electron microscopy at CSA=2.0 mrad

EMDB-41805: 
Cryo-EM structure of murine Thrombopoietin receptor ectodomain in complex with Tpo

PDB-8u18: 
Cryo-EM structure of murine Thrombopoietin receptor ectodomain in complex with Tpo
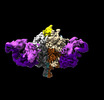
EMDB-41048: 
Lassa GPC Trimer in complex with Fab 8.11G and nanobody D5

PDB-8t5c: 
Lassa GPC Trimer in complex with Fab 8.11G and nanobody D5

EMDB-29447: 
Sec39:Use1:Sec20:Tip20 Local Refinement of the Dsl1:Qb:Qc Complex #2

EMDB-29292: 
HIV-1 envelope trimer bound to one CD4 molecule on membranes

EMDB-29293: 
HIV-1 envelope trimer bound to two CD4 molecules on membranes

EMDB-29294: 
HIV-1 envelope trimer bound to three CD4 molecules on membranes

EMDB-29295: 
Consensus structure of HIV-1 envelope trimer bound to CD4 receptors on membranes

EMDB-41045: 
Unliganded HIV-1 Env on virion

EMDB-28760: 
Dsl1:Sec39 Local Refinement of the Dsl1:Qb:Qc Complex

EMDB-28762: 
Dsl1:Tip20 Local Refinement of the Dsl1:Qb:Qc Complex

EMDB-28768: 
Sec39:Use1:Sec20:Tip20 Local Refinement of the Dsl1:Qb:Qc Complex

EMDB-28774: 
Consensus Map of the Dsl1:Qb:Qc Complex

EMDB-41308: 
Tetrahymena Ribozyme scaffolded Zika Virus xrRNA

EMDB-41311: 
Tetrahymena Ribozyme scaffolded TABV xrRNA

EMDB-41312: 
Tetrahymena Ribozyme scaffolded Fluoride riboswitch

EMDB-41313: 
Tetrahymena Ribozyme cryo-EM scaffold

PDB-8tjq: 
Tetrahymena Ribozyme scaffolded Zika Virus xrRNA

PDB-8tju: 
Tetrahymena Ribozyme scaffolded TABV xrRNA

PDB-8tjv: 
Tetrahymena Ribozyme scaffolded Fluoride riboswitch

PDB-8tjx: 
Tetrahymena Ribozyme cryo-EM scaffold
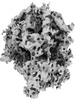
EMDB-41205: 
Cryo-electron tomography reconstruction of mammalian 80S ribosome in the non-rotated state with P, A/T-site tRNA, and eEF1A cofactor from EMPIAR-10064 dataset by constrained classification

EMDB-41207: 
Cryo-electron tomography reconstruction of mammalian 80S ribosome from EMPIAR-10064 in the translocation intermediates (TI) state with eEF2 cofactor and closed L1 by constrained classification.

EMDB-41210: 
Cryo-electron tomography reconstruction of mammalian 80S ribosome in the translocation intermediates (TI) state with eEF2 cofactor and open L1 from EMPIAR-10064 dataset by constrained classification.

EMDB-41211: 
Cryo-electron tomography reconstruction of mammalian 80S ribosome in the non-rotated state with P, A/T-site tRNA from EMPIAR-10064 dataset by constrained classification.

EMDB-41212: 
Cryo-electron tomography reconstruction of mammalian 80S ribosome in the non-rotated state with E, P, A/T-site tRNA and eEF1A cofactor from EMPIAR-10064 dataset by constrained classification.

EMDB-41220: 
Cryo-electron tomography reconstruction of M. pneumoniae 70S ribosome in decoding state with EF-Tu-tRNA and P-site tRNA from EMPIAR-10499 dataset by constrained classification.

EMDB-41221: 
Cryo-electron tomography reconstruction of M. pneumoniae 70S ribosome in classical non-rotated state with L1 open, obtained from EMPIAR-10499 dataset by constrained classification

EMDB-41222: 
Cryo-electron tomography reconstruction of M. pneumoniae 70S ribosome in the classical non-rotated state with half-closed L1 stalk, obtained from EMPIAR-10499 dataset by constrained classification

EMDB-41223: 
Cryo-electron tomography reconstruction of 80S ribosome in non-rotated state class 1, obtained from the EMPIAR-10987 dataset by constrained 3D classification.

EMDB-41224: 
Cryo-electron tomography reconstruction of 80S ribosome in non-rotated state class 2, obtained from the EMPIAR-10987 dataset by constrained 3D classification.

EMDB-41225: 
Cryo-electron tomography reconstruction of 80S ribosome in non-rotated state class 3, obtained from the EMPIAR-10987 dataset by constrained 3D classification.
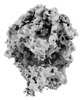
EMDB-41226: 
Cryo-electron tomography reconstruction of 80S ribosome in non-rotated decoding state class 4, obtained from the EMPIAR-10987 dataset by constrained 3D classification.

EMDB-41227: 
Cryo-electron tomography reconstruction of 80S ribosome in Translocation-intermediate-POST-3 state class 5, obtained from the EMPIAR-10987 dataset by constrained 3D classification.

EMDB-41196: 
A 3.2 Angstrom structure of HIV-1 CA-SP1 from 5 tomograms in EMPIAR-10164 dataset by single-particle cryo-electron tomography

EMDB-41197: 
A 3 Angstrom structure of HIV-1 CA-SP1 from EMPIAR-10164 dataset by single-particle cryo-electron tomography

EMDB-41199: 
1.8 Angstrom mouse heavy chain apoferritin from EMPIAR-11273 dataset by single-particle cryo-electron tomography

EMDB-28204: 
CryoEM structure of the Dsl1 complex bound to SNAREs Sec20 and Use1

PDB-8eki: 
CryoEM structure of the Dsl1 complex bound to SNAREs Sec20 and Use1

EMDB-28153: 
cryo-EM structure of TMEM63A in nanodisc

EMDB-28154: 
cryo-EM structure of TMEM63B in LMNG

PDB-8ehw: 
cryo-EM structure of TMEM63A in nanodisc

PDB-8ehx: 
cryo-EM structure of TMEM63B in LMNG

EMDB-41302: 
Lassa GPC trimer in complex with Fab GP23

EMDB-27781: 
Cryo-EM structure of 227 Fab in complex with (NPNA)8 peptide

EMDB-27784: 
Cryo-EM structure of 239 Fab in complex with recombinant shortened Plasmodium falciparum circumsporozoite protein (rsCSP)

EMDB-27785: 
Cryo-EM structure of 311 Fab in complex with recombinant shortened Plasmodium falciparum circumsporozoite protein (rsCSP)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
