-検索条件
-検索結果
検索 (著者・登録者: gustavsson & e)の結果全45件を表示しています

EMDB-17014: 
Consensus map of HSV-1 DNA polymerase-processivity factor complex in pre-translocation state

EMDB-17013: 
HSV-1 DNA polymerase-processivity factor complex in halted elongation state consensus map

EMDB-17018: 
Consensus map of HSV-1 DNA polymerase-processivity factor complex in exonuclease state

EMDB-16918: 
Focused refinement map of HSV-1 DNA polymerase in pre-translocation state

EMDB-16919: 
Focused refinement map of HSV-1 DNA polymerase processivity factor in pre-translocation state

EMDB-16924: 
Focused refinement of HSV-1 DNA polymerase in halted elongation state

EMDB-16925: 
Focused refinement of HSV-1 DNA polymerase processivity factor in halted elongation state

EMDB-16927: 
Focused refinement map of HSV-1 DNA polymerase in exonuclease state

EMDB-16928: 
Focused refinement map of HSV-1 DNA polymerase processivity factor in exonuclease state

EMDB-16906: 
HSV-1 DNA polymerase-processivity factor complex in pre-translocation state

EMDB-16907: 
HSV-1 DNA polymerase-processivity factor complex in halted elongation state

EMDB-16909: 
HSV-1 DNA polymerase-processivity factor complex in exonuclease state

EMDB-16910: 
HSV-1 DNA polymerase-processivity factor complex in exonuclease state active site

EMDB-16911: 
HSV-1 DNA polymerase active site in alternative exonuclease state

EMDB-16912: 
HSV-1 DNA polymerase beta-hairpin loop

PDB-8oj6: 
HSV-1 DNA polymerase-processivity factor complex in pre-translocation state

PDB-8oj7: 
HSV-1 DNA polymerase-processivity factor complex in halted elongation state

PDB-8oja: 
HSV-1 DNA polymerase-processivity factor complex in exonuclease state

PDB-8ojb: 
HSV-1 DNA polymerase-processivity factor complex in exonuclease state active site

PDB-8ojc: 
HSV-1 DNA polymerase active site in alternative exonuclease state

PDB-8ojd: 
HSV-1 DNA polymerase beta-hairpin loop

EMDB-14438: 
VAR2 complex with PAM1.4

EMDB-14446: 
VAR2CSA APO

PDB-7z12: 
VAR2 complex with PAM1.4

PDB-7z1h: 
VAR2CSA APO
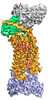
EMDB-25171: 
Cryo-EM structure of ACKR3 in complex with CXCL12, an intracellular Fab, and an extracellular Fab

EMDB-25172: 
Cryo-EM structure of ACKR3 in complex with chemokine N-terminal mutant CXCL12_LRHQ, an intracellular Fab, and an extracellular Fab
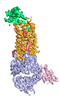
EMDB-25173: 
Cryo-EM structure of ACKR3 in complex with CXCL12 and an intracellular Fab
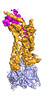
EMDB-25174: 
Cryo-EM structure of human ACKR3 in complex with chemokine N-terminal mutant CXCL12_LRHQ and an intracellular Fab
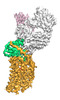
EMDB-25175: 
Cryo-EM structure of human ACKR3 in complex with CXCL12, a small molecule partial agonist CCX662, and an extracellular Fab
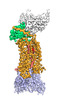
EMDB-25176: 
Cryo-EM structure of human ACKR3 in complex with CXCL12, a small molecule partial agonist CCX662, an extracellular Fab, and an intracellular Fab
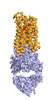
EMDB-25177: 
Cryo-EM structure of human ACKR3 in complex with a small molecule partial agonist CCX662, and an intracellular Fab

PDB-7sk3: 
Cryo-EM structure of ACKR3 in complex with CXCL12, an intracellular Fab, and an extracellular Fab

PDB-7sk4: 
Cryo-EM structure of ACKR3 in complex with chemokine N-terminal mutant CXCL12_LRHQ, an intracellular Fab, and an extracellular Fab

PDB-7sk5: 
Cryo-EM structure of ACKR3 in complex with CXCL12 and an intracellular Fab

PDB-7sk6: 
Cryo-EM structure of human ACKR3 in complex with chemokine N-terminal mutant CXCL12_LRHQ and an intracellular Fab

PDB-7sk7: 
Cryo-EM structure of human ACKR3 in complex with CXCL12, a small molecule partial agonist CCX662, and an extracellular Fab

PDB-7sk8: 
Cryo-EM structure of human ACKR3 in complex with CXCL12, a small molecule partial agonist CCX662, an extracellular Fab, and an intracellular Fab

PDB-7sk9: 
Cryo-EM structure of human ACKR3 in complex with a small molecule partial agonist CCX662, and an intracellular Fab

EMDB-12018: 
VAR2CSA full ectodomain in present of plCS, DBL1-DBL4

EMDB-12477: 
Cryo-EM structure of VAR2CSA FCR3 domain DBL5/6

PDB-7b54: 
VAR2CSA full ectodomain in present of plCS, DBL1-DBL4

PDB-7nnh: 
Cryo-EM structure of VAR2CSA FCR3 domain DBL5/6

EMDB-12017: 
VAR2CSA full ectodomain

PDB-7b52: 
VAR2CSA full ectodomain
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
