-検索条件
-検索結果
検索 (データベース: PDB)の結果21,961件中、1から50件目までを表示しています

PDB-8rjf: 
TadA/CpaF with ADP

PDB-8rkd: 
TadA/CpaF with AMPPNP

PDB-8rkl: 
TadA/CpaF nucleotide free

PDB-9cip: 
MicroED structure of the C11 cysteine protease clostripain

PDB-8xit: 
Cryo-EM structure of sheep VMAT2 dimer in an atypical fold

PDB-8xiu: 
Cryo-EM structure of a frog VMAT2 in an apo conformation

PDB-8qro: 
ASCT2 trimer in lipid nanodiscs with bound glutamine and Na+ ions in the outward-facing state (OFS)

PDB-8qrp: 
ASCT2 protomer in lipid nanodiscs with bound glutamine and Na+ ions in the outward-facing state (OFS.1)

PDB-8qrq: 
ASCT2 protomer in lipid nanodiscs with bound glutamine and Na+ ions in the outward-facing state (OFS.2)

PDB-8qrr: 
ASCT2 protomer in lipid nanodiscs with bound glutamine and Na+ ions in the outward-facing state (OFS.3)

PDB-8qrs: 
ASCT2 protomer in lipid nanodiscs with bound glutamine and Na+ ions in the intermediate outward-facing state (iOFS-up)

PDB-8qru: 
ASCT2 protomer in lipid nanodiscs with bound glutamine and Na+ ions in the intermediate outward-facing state (iOFS-down)

PDB-8qrv: 
ASCT2 protomer in lipid nanodiscs under low Na+ concentration in the outward-facing state (OFS)

PDB-8qrw: 
ASCT2 protomer in lipid nanodiscs under low Na+ concentration in the intermediate outward-facing state (iOFS-up)

PDB-8y6v: 
Near-atomic structure of icosahedrally averaged jumbo bacteriophage PhiKZ capsid

PDB-8wix: 
cryo-EM structure of alligator haemoglobin in carbonmonoxy form

PDB-8wiy: 
cryo-EM structure of alligator haemoglobin in oxy form

PDB-8wiz: 
cryo-EM structure of alligator haemoglobin in deoxy form

PDB-8wj0: 
cryo-EM structure of human haemoglobin in carbonmonoxy form

PDB-8wj1: 
cryo-EM structure of human haemoglobin in oxy form

PDB-8wj2: 
cryo-EM structure of human haemoglobin in deoxy form

PDB-9c47: 
TRRAP module of the human TIP60 complex

PDB-9c4b: 
Second BAF53a of the human TIP60 complex

PDB-8yf6: 
Cryo-EM structure of Dragon Grouper nervous necrosis virus-like particle at pH8.0 (3.23A)

PDB-8yf7: 
Cryo-EM structure of Dragon Grouper nervous necrosis virus-like particle at pH6.5 (2.82A)

PDB-8yf8: 
Cryo-EM structure of Dragon Grouper nervous necrosis virus-like particle at pH5.0 (3.52A)

PDB-8yf9: 
Cryo-EM structure of Dragon Grouper nervous necrosis virion at pH6.5 (3.12A)

PDB-8xvb: 
Cryo-EM structure of ATP-DNA-MuB filaments

PDB-8xvc: 
CryoEM structure of ADP-DNA-MuB conformation1

PDB-8xvd: 
CryoEM structure of ADP-DNA-MuB conformation2

PDB-8tzv: 
Apo form of human ATE1

PDB-8uau: 
human ATE1 in complex with Arg-tRNA and a peptide substrate

PDB-8ykf: 
The DSR2-DSAD1 complex with DSAD1 on the opposite sides

PDB-8yl5: 
The DSR2-DSAD1 complex with DSAD1 on the same sides

PDB-8yln: 
The structure of DSR2-Tail tube complex

PDB-8ylt: 
The structure of DSR2 and NAD+ complex

PDB-8z18: 
The tetramer complex of DSR2 and tube-forming domain of phage tail tube protein

PDB-8ztr: 
The dimer complex of DSR2 and tube-forming domain of phage tail tube protein

PDB-8xfb: 
Cryo-EM structure of partial dimeric WDR11-FAM91A1 complex

PDB-8z9m: 
Cryo-EM structure of dimeric WDR11-FAM91A1 complex

PDB-9ash: 
Cryo-EM structure of the active Lactococcus lactis Csm bound to target in post-cleavage stage

PDB-9asi: 
Cryo-EM structure of the active Lactococcus lactis Csm bound to target in pre-cleavage stage

PDB-8ofi: 
Ivabradine bound to HCN4 channel
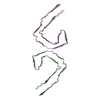
PDB-9bxi: 
Paired Helical Filament of tau amyloids found in Down Syndrome individuals
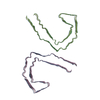
PDB-9bxo: 
Straight Filament of tau amyloids found in Down Syndrome individuals
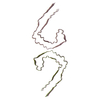
PDB-9bxq: 
Paired Helical Filaments purified from Down Syndrome individual brain tissue applied to graphene oxide antibody affinity grids

PDB-9bxr: 
Straight Filaments purified from Down Syndrome individual brain tissue applied to graphene oxide antibody affinity grids

PDB-8u39: 
Structure of Human Mitochondrial Chaperonin V72I mutant

PDB-8kdm: 
Structure of SARS-CoV Spike protein complexed with antibody PW5-5

PDB-8kdr: 
The local refined map of SARS-CoV-2 XBB Variant Spike protein complexed with antibody PW5-535
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
