+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1965 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structural and Functional Studies of LRP6 Ectodomain Reveal a Platform for Wnt Signaling | |||||||||
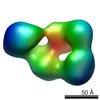
 マップデータ マップデータ | This is a surface rendering of the ectodomain of LRP6 bound to its chaperone Mesd | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | LDL-receptor-related protein 6 /  Wnt signaling pathway (Wntシグナル経路) / Wnt co-receptor Wnt signaling pathway (Wntシグナル経路) / Wnt co-receptor | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  ネガティブ染色法 / 解像度: 26.0 Å ネガティブ染色法 / 解像度: 26.0 Å | |||||||||
 データ登録者 データ登録者 | Chen S / Bubeck D / MacDonald BT / Liang WX / Mao JH / Malinauskas T / Llorca O / Aricescu AR / Siebold C / He X / Jones EY | |||||||||
 引用 引用 |  ジャーナル: Dev Cell / 年: 2011 ジャーナル: Dev Cell / 年: 2011タイトル: Structural and functional studies of LRP6 ectodomain reveal a platform for Wnt signaling. 著者: Shuo Chen / Doryen Bubeck / Bryan T MacDonald / Wen-Xue Liang / Jian-Hua Mao / Tomas Malinauskas / Oscar Llorca / A Radu Aricescu / Christian Siebold / Xi He / E Yvonne Jones /  要旨: LDL-receptor-related protein 6 (LRP6), alongside Frizzled receptors, transduces Wnt signaling across the plasma membrane. The LRP6 ectodomain comprises four tandem β-propeller-EGF-like domain (PE) ...LDL-receptor-related protein 6 (LRP6), alongside Frizzled receptors, transduces Wnt signaling across the plasma membrane. The LRP6 ectodomain comprises four tandem β-propeller-EGF-like domain (PE) pairs that harbor binding sites for Wnt morphogens and their antagonists including Dickkopf 1 (Dkk1). To understand how these multiple interactions are integrated, we combined crystallographic analysis of the third and fourth PE pairs with electron microscopy (EM) to determine the complete ectodomain structure. An extensive inter-pair interface, conserved for the first-to-second and third-to-fourth PE interactions, contributes to a compact platform-like architecture, which is disrupted by mutations implicated in developmental diseases. EM reconstruction of the LRP6 platform bound to chaperone Mesd exemplifies a binding mode spanning PE pairs. Cellular and binding assays identify overlapping Wnt3a- and Dkk1-binding surfaces on the third PE pair, consistent with steric competition, but also suggest a model in which the platform structure supports an interplay of ligands through multiple interaction sites. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1965.map.gz emd_1965.map.gz | 790.7 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1965-v30.xml emd-1965-v30.xml emd-1965.xml emd-1965.xml | 9.7 KB 9.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1965_LRP6_MESD3.jpg 1965_LRP6_MESD3.jpg | 42.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1965 http://ftp.pdbj.org/pub/emdb/structures/EMD-1965 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1965 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1965 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1965.map.gz / 形式: CCP4 / 大きさ: 825.2 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1965.map.gz / 形式: CCP4 / 大きさ: 825.2 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is a surface rendering of the ectodomain of LRP6 bound to its chaperone Mesd | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.56 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Ectodomain of LRP6 residues 20 to 1361 and chaperone Mesd
| 全体 | 名称: Ectodomain of LRP6 residues 20 to 1361 and chaperone Mesd |
|---|---|
| 要素 |
|
-超分子 #1000: Ectodomain of LRP6 residues 20 to 1361 and chaperone Mesd
| 超分子 | 名称: Ectodomain of LRP6 residues 20 to 1361 and chaperone Mesd タイプ: sample / ID: 1000 / 詳細: The sample was monodisperse / 集合状態: one LRP6 binds to one Mesd / Number unique components: 2 |
|---|---|
| 分子量 | 実験値: 200 KDa / 手法: Multi angle light scattering |
-分子 #1: LDL-receptor-related protein 6
| 分子 | 名称: LDL-receptor-related protein 6 / タイプ: protein_or_peptide / ID: 1 / Name.synonym: LRP6 / コピー数: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 実験値: 100 KDa |
| 組換発現 | 生物種: HEK293S / 組換プラスミド: pHLsec vector |
-分子 #2: Mesoderm development protein
| 分子 | 名称: Mesoderm development protein / タイプ: protein_or_peptide / ID: 2 / Name.synonym: Mesd / コピー数: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 実験値: 100 KDa |
| 組換発現 | 生物種: HEK293S / 組換プラスミド: pHLsec vector |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法 ネガティブ染色法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.015 mg/mL |
|---|---|
| 緩衝液 | pH: 8 / 詳細: 150 mM NaCl,10 mM Tris-HCl |
| 染色 | タイプ: NEGATIVE 詳細: Grids were negatively stained with 0.75% uranyl formate using the two-drop method |
| グリッド | 詳細: carbon-coated copper grids |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 1230 |
|---|---|
| 電子線 | 加速電圧: 100 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.9 mm / 倍率(公称値): 72500 Bright-field microscopy / Cs: 2.9 mm / 倍率(公称値): 72500 |
| 試料ステージ | 試料ホルダー: single-tilt / 試料ホルダーモデル: JEOL |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: TVIPS TEMCAM-F416 (4k x 4k) デジタル化 - サンプリング間隔: 4.56 µm / 平均電子線量: 10 e/Å2 |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 26.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMAN, XMIPP / 使用した粒子像数: 7928 |
|---|
 ムービー
ムービー コントローラー
コントローラー