+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of HACE1 dimer | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  E3 ubiquitin ligase (ユビキチンリガーゼ) / E3 ubiquitin ligase (ユビキチンリガーゼ) /  tumor suppressor (がん抑制遺伝子) / Post-translational modifier / tumor suppressor (がん抑制遺伝子) / Post-translational modifier /  Protein degradation (タンパク質分解) / Protein degradation (タンパク質分解) /  ANTITUMOR PROTEIN ANTITUMOR PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報HECT-type E3 ubiquitin transferase / Golgi cisterna membrane / Rac protein signal transduction / Golgi organization / protein K48-linked ubiquitination /  regulation of cell migration / regulation of cell migration /  small GTPase binding / protein polyubiquitination / ubiquitin-protein transferase activity / small GTPase binding / protein polyubiquitination / ubiquitin-protein transferase activity /  ubiquitin protein ligase activity ...HECT-type E3 ubiquitin transferase / Golgi cisterna membrane / Rac protein signal transduction / Golgi organization / protein K48-linked ubiquitination / ubiquitin protein ligase activity ...HECT-type E3 ubiquitin transferase / Golgi cisterna membrane / Rac protein signal transduction / Golgi organization / protein K48-linked ubiquitination /  regulation of cell migration / regulation of cell migration /  small GTPase binding / protein polyubiquitination / ubiquitin-protein transferase activity / small GTPase binding / protein polyubiquitination / ubiquitin-protein transferase activity /  ubiquitin protein ligase activity / Antigen processing: Ubiquitination & Proteasome degradation / ubiquitin-dependent protein catabolic process / ubiquitin protein ligase activity / Antigen processing: Ubiquitination & Proteasome degradation / ubiquitin-dependent protein catabolic process /  membrane fusion / protein ubiquitination / membrane fusion / protein ubiquitination /  nuclear body / nuclear body /  細胞周期 / 細胞周期 /  ゴルジ体 / ゴルジ体 /  小胞体 / 小胞体 /  細胞核 / 細胞核 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
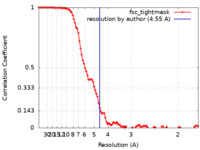
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.55 Å クライオ電子顕微鏡法 / 解像度: 4.55 Å | |||||||||
 データ登録者 データ登録者 | Singh S / Machida S / Tulsian NK / Choong YK / Ng J / Shanker S / Yaochen LD / Shi J / Sivaraman J | |||||||||
| 資金援助 |  シンガポール, 1件 シンガポール, 1件
| |||||||||
 引用 引用 |  ジャーナル: Adv Sci (Weinh) / 年: 2023 ジャーナル: Adv Sci (Weinh) / 年: 2023タイトル: Structural Basis for the Enzymatic Activity of the HACE1 HECT-Type E3 Ligase Through N-Terminal Helix Dimerization. 著者: Sunil Singh / Satoru Machida / Nikhil Kumar Tulsian / Yeu Khai Choong / Joel Ng / Srihari Shankar / Yaochen Liu / Krisha Vashdev Chandiramani / Jian Shi / J Sivaraman /  要旨: HACE1 is an ankyrin repeat (AKR) containing HECT-type E3 ubiquitin ligase that interacts with and ubiquitinates multiple substrates. While HACE1 is a well-known tumor suppressor, its structure and ...HACE1 is an ankyrin repeat (AKR) containing HECT-type E3 ubiquitin ligase that interacts with and ubiquitinates multiple substrates. While HACE1 is a well-known tumor suppressor, its structure and mode of ubiquitination are not understood. The authors present the cryo-EM structures of human HACE1 along with in vitro functional studies that provide insights into how the enzymatic activity of HACE1 is regulated. HACE1 comprises of an N-terminal AKR domain, a middle (MID) domain, and a C-terminal HECT domain. Its unique G-shaped architecture interacts as a homodimer, with monomers arranged in an antiparallel manner. In this dimeric arrangement, HACE1 ubiquitination activity is hampered, as the N-terminal helix of one monomer restricts access to the C-terminal domain of the other. The in vitro ubiquitination assays, hydrogen-deuterium exchange mass spectrometry (HDX-MS) analysis, mutagenesis, and in silico modeling suggest that the HACE1 MID domain plays a crucial role along with the AKRs in RAC1 substrate recognition. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34586.map.gz emd_34586.map.gz | 84 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34586-v30.xml emd-34586-v30.xml emd-34586.xml emd-34586.xml | 15.5 KB 15.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_34586_fsc.xml emd_34586_fsc.xml | 10.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_34586.png emd_34586.png | 84.8 KB | ||
| マスクデータ |  emd_34586_msk_1.map emd_34586_msk_1.map | 91.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-34586.cif.gz emd-34586.cif.gz | 6.2 KB | ||
| その他 |  emd_34586_half_map_1.map.gz emd_34586_half_map_1.map.gz emd_34586_half_map_2.map.gz emd_34586_half_map_2.map.gz | 84.6 MB 84.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34586 http://ftp.pdbj.org/pub/emdb/structures/EMD-34586 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34586 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34586 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8haeMC  8h8xC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34586.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34586.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.8584 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_34586_msk_1.map emd_34586_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
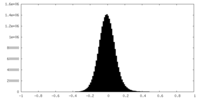
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_34586_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
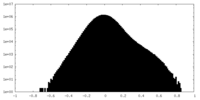
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_34586_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : HACE1 dimer
| 全体 | 名称: HACE1 dimer |
|---|---|
| 要素 |
|
-超分子 #1: HACE1 dimer
| 超分子 | 名称: HACE1 dimer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 200 KDa |
-分子 #1: E3 ubiquitin-protein ligase HACE1
| 分子 | 名称: E3 ubiquitin-protein ligase HACE1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: HECT-type E3 ubiquitin transferase |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 102.449672 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21 (大腸菌) Escherichia coli BL21 (大腸菌) |
| 配列 | 文字列: MERAMEQLNR LTRSLRRART VELPEDNETA VYTLMPMVMA DQHRSVSELL SNSKFDVNYA FGRVKRSLLH IAANCGSVEC LVLLLKKGA NPNYQDISGC TPLHLAARNG QKKCMSKLLE YSADVNICNN EGLTAIHWLA VNGRTELLHD LVQHVSDVDV E DAMGQTAL ...文字列: MERAMEQLNR LTRSLRRART VELPEDNETA VYTLMPMVMA DQHRSVSELL SNSKFDVNYA FGRVKRSLLH IAANCGSVEC LVLLLKKGA NPNYQDISGC TPLHLAARNG QKKCMSKLLE YSADVNICNN EGLTAIHWLA VNGRTELLHD LVQHVSDVDV E DAMGQTAL HVACQNGHKT TVQCLLDSGA DINRPNVSGA TPLYFACSHG QRDTAQILLL RGAKYLPDKN GVTPLDLCVQ GG YGETCEV LIQYHPRLFQ TIIQMTQNED LRENMLRQVL EHLSQQSESQ YLKILTSLAE VATTNGHKLL SLSSNYDAQM KSL LRIVRM FCHVFRIGPS SPSNGIDMGY NGNKTPRSQV FKPLELLWHS LDEWLVLIAT ELMKNKRDST EITSILLKQK GQDQ DAASI PPFEPPGPGS YENLSTGTRE SKPDALAGRQ EASADCQDVI SMTANRLSAV IQAFYMCCSC QMPPGMTSPR FIEFV CKHD EVLKCFVNRN PKIIFDHFHF LLECPELMSR FMHIIKAQPF KDRCEWFYEH LHSGQPDSDM VHRPVNENDI LLVHRD SIF RSSCEVVSKA NCAKLKQGIA VRFHGEEGMG QGVVREWFDI LSNEIVNPDY ALFTQSADGT TFQPNSNSYV NPDHLNY FR FAGQILGLAL NHRQLVNIYF TRSFYKHILG IPVNYQDVAS IDPEYAKNLQ WILDNDISDL GLELTFSVET DVFGAMEE V PLKPGGGSIL VTQNNKAEYV QLVTELRMTR AIQPQINAFL QGFHMFIPPS LIQLFDEYEL ELLLSGMPEI DVSDWIKNT EYTSGYERED PVIQWFWEVV EDITQEERVL LLQFVTGSSR VPHGGFANIM GGSGLQNFTI AAVPYTPNLL PTSSTCINML KLPEYPSKE ILKDRLLVAL HCGSYGYTMA UniProtKB: E3 ubiquitin-protein ligase HACE1 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 200 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 120 sec. |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 0.5 µm Bright-field microscopy / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
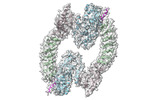
| 得られたモデル |  PDB-8hae: |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X