+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21536 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CaMKII alpha-30 Cryo-EM reconstruction - Class B | |||||||||
 マップデータ マップデータ | Primary Map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Calcium calmodulin dependent protein kinase II /  holoenzyme (酵素) / holoenzyme (酵素) /  splicing / splicing /  SIGNALING PROTEIN / SIGNALING PROTEIN /  SIGNALING PROTEIN-TRANSFERASE complex SIGNALING PROTEIN-TRANSFERASE complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報peptidyl-threonine autophosphorylation / regulation of endocannabinoid signaling pathway / calcium- and calmodulin-dependent protein kinase complex /  regulation of neuron migration / regulation of neuron migration /  Ca2+/calmodulin-dependent protein kinase / regulation of neurotransmitter secretion / dendritic spine development / positive regulation of calcium ion transport / Trafficking of AMPA receptors / negative regulation of hydrolase activity ...peptidyl-threonine autophosphorylation / regulation of endocannabinoid signaling pathway / calcium- and calmodulin-dependent protein kinase complex / Ca2+/calmodulin-dependent protein kinase / regulation of neurotransmitter secretion / dendritic spine development / positive regulation of calcium ion transport / Trafficking of AMPA receptors / negative regulation of hydrolase activity ...peptidyl-threonine autophosphorylation / regulation of endocannabinoid signaling pathway / calcium- and calmodulin-dependent protein kinase complex /  regulation of neuron migration / regulation of neuron migration /  Ca2+/calmodulin-dependent protein kinase / regulation of neurotransmitter secretion / dendritic spine development / positive regulation of calcium ion transport / Trafficking of AMPA receptors / negative regulation of hydrolase activity / Assembly and cell surface presentation of NMDA receptors / calmodulin-dependent protein kinase activity / regulation of mitochondrial membrane permeability involved in apoptotic process / CaMK IV-mediated phosphorylation of CREB / Negative regulation of NMDA receptor-mediated neuronal transmission / positive regulation of cardiac muscle cell apoptotic process / Unblocking of NMDA receptors, glutamate binding and activation / regulation of neuronal synaptic plasticity / Phase 0 - rapid depolarisation / Ion transport by P-type ATPases / Ca2+/calmodulin-dependent protein kinase / regulation of neurotransmitter secretion / dendritic spine development / positive regulation of calcium ion transport / Trafficking of AMPA receptors / negative regulation of hydrolase activity / Assembly and cell surface presentation of NMDA receptors / calmodulin-dependent protein kinase activity / regulation of mitochondrial membrane permeability involved in apoptotic process / CaMK IV-mediated phosphorylation of CREB / Negative regulation of NMDA receptor-mediated neuronal transmission / positive regulation of cardiac muscle cell apoptotic process / Unblocking of NMDA receptors, glutamate binding and activation / regulation of neuronal synaptic plasticity / Phase 0 - rapid depolarisation / Ion transport by P-type ATPases /  長期増強 / Regulation of MECP2 expression and activity / HSF1-dependent transactivation / 長期増強 / Regulation of MECP2 expression and activity / HSF1-dependent transactivation /  glutamate receptor binding / cellular response to interferon-beta / Ion homeostasis / Ras activation upon Ca2+ influx through NMDA receptor / response to ischemia / angiotensin-activated signaling pathway / G1/S transition of mitotic cell cycle / RAF activation / positive regulation of receptor signaling pathway via JAK-STAT / cellular response to type II interferon / Signaling by RAF1 mutants / endocytic vesicle membrane / Signaling by moderate kinase activity BRAF mutants / Paradoxical activation of RAF signaling by kinase inactive BRAF / Signaling downstream of RAS mutants / calcium ion transport / Interferon gamma signaling / Signaling by BRAF and RAF1 fusions / Ca2+ pathway / positive regulation of NF-kappaB transcription factor activity / glutamate receptor binding / cellular response to interferon-beta / Ion homeostasis / Ras activation upon Ca2+ influx through NMDA receptor / response to ischemia / angiotensin-activated signaling pathway / G1/S transition of mitotic cell cycle / RAF activation / positive regulation of receptor signaling pathway via JAK-STAT / cellular response to type II interferon / Signaling by RAF1 mutants / endocytic vesicle membrane / Signaling by moderate kinase activity BRAF mutants / Paradoxical activation of RAF signaling by kinase inactive BRAF / Signaling downstream of RAS mutants / calcium ion transport / Interferon gamma signaling / Signaling by BRAF and RAF1 fusions / Ca2+ pathway / positive regulation of NF-kappaB transcription factor activity /  kinase activity / RAF/MAP kinase cascade / peptidyl-serine phosphorylation / kinase activity / RAF/MAP kinase cascade / peptidyl-serine phosphorylation /  樹状突起スパイン / 樹状突起スパイン /  postsynaptic density / protein autophosphorylation / postsynaptic density / protein autophosphorylation /  calmodulin binding / neuron projection / calmodulin binding / neuron projection /  protein phosphorylation / protein serine kinase activity / protein serine/threonine kinase activity / protein homodimerization activity / protein phosphorylation / protein serine kinase activity / protein serine/threonine kinase activity / protein homodimerization activity /  ミトコンドリア / ミトコンドリア /  核質 / 核質 /  ATP binding / identical protein binding / ATP binding / identical protein binding /  metal ion binding / metal ion binding /  細胞核 / 細胞核 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
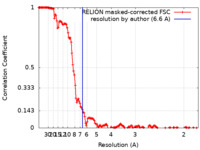
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 6.6 Å クライオ電子顕微鏡法 / 解像度: 6.6 Å | |||||||||
 データ登録者 データ登録者 | Chao LH / Stratton MM | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Signal / 年: 2020 ジャーナル: Sci Signal / 年: 2020タイトル: Heterogeneity in human hippocampal CaMKII transcripts reveals allosteric hub-dependent regulation. 著者: Roman Sloutsky / Noelle Dziedzic / Matthew J Dunn / Rachel M Bates / Ana P Torres-Ocampo / Sivakumar Boopathy / Brendan Page / John G Weeks / Luke H Chao / Margaret M Stratton /  要旨: Calcium/calmodulin-dependent protein kinase II (CaMKII) plays a central role in Ca signaling throughout the body. In the hippocampus, CaMKII is required for learning and memory. Vertebrate genomes ...Calcium/calmodulin-dependent protein kinase II (CaMKII) plays a central role in Ca signaling throughout the body. In the hippocampus, CaMKII is required for learning and memory. Vertebrate genomes encode four CaMKII homologs: CaMKIIα, CaMKIIβ, CaMKIIγ, and CaMKIIδ. All CaMKIIs consist of a kinase domain, a regulatory segment, a variable linker region, and a hub domain, which is responsible for oligomerization. The four proteins differ primarily in linker length and composition because of extensive alternative splicing. Here, we report the heterogeneity of CaMKII transcripts in three complex samples of human hippocampus using deep sequencing. We showed that hippocampal cells contain a diverse collection of over 70 CaMKII transcripts from all four CaMKII-encoding genes. We characterized the Ca/CaM sensitivity of hippocampal CaMKII variants spanning a broad range of linker lengths and compositions. The effect of the variable linker on Ca/CaM sensitivity depended on the kinase and hub domains. Moreover, we revealed a previously uncharacterized role for the hub domain as an allosteric regulator of kinase activity, which may provide a pharmacological target for modulating CaMKII activity. Using small-angle x-ray scattering and single-particle cryo-electron microscopy (cryo-EM), we present evidence for extensive interactions between the kinase and the hub domains, even in the presence of a 30-residue linker. Together, these data suggest that Ca/CaM sensitivity in CaMKII is homolog dependent and includes substantial contributions from the hub domain. Our sequencing approach, combined with biochemistry, provides insights into understanding the complex pool of endogenous CaMKII splice variants. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21536.map.gz emd_21536.map.gz | 224.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21536-v30.xml emd-21536-v30.xml emd-21536.xml emd-21536.xml | 18 KB 18 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_21536_fsc.xml emd_21536_fsc.xml | 14.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
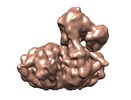
| 画像 |  emd_21536.png emd_21536.png | 193.4 KB | ||
| マスクデータ |  emd_21536_msk_1.map emd_21536_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-21536.cif.gz emd-21536.cif.gz | 5.9 KB | ||
| その他 |  emd_21536_half_map_1.map.gz emd_21536_half_map_1.map.gz emd_21536_half_map_2.map.gz emd_21536_half_map_2.map.gz | 193.6 MB 193.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21536 http://ftp.pdbj.org/pub/emdb/structures/EMD-21536 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21536 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21536 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21536.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21536.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Primary Map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.91 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_21536_msk_1.map emd_21536_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
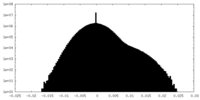
| 密度ヒストグラム |
-ハーフマップ: half-map 1
| ファイル | emd_21536_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half-map 2
| ファイル | emd_21536_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CaMKII alpha-30
| 全体 | 名称: CaMKII alpha-30 |
|---|---|
| 要素 |
|
-超分子 #1: CaMKII alpha-30
| 超分子 | 名称: CaMKII alpha-30 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Calcium/calmodulin-dependent protein kinase type II subunit alpha
| 分子 | 名称: Calcium/calmodulin-dependent protein kinase type II subunit alpha タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO / EC番号:  Ca2+/calmodulin-dependent protein kinase Ca2+/calmodulin-dependent protein kinase |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 53.633953 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: STRFTEEYQL FEELGKGAFS VVRRCVKVLA GQEYAAKIIN TKKLSARDHQ KLEREARICR LLKHPNIVRL HDSISEEGHH YLIFDLVTG GELFEDIVAR EYYSEADASH CIQQILEAVL HCHQMGVVHR DLKPENLLLA SKLKGAAVKL ADFGLAIEVE G EQQAWFGF ...文字列: STRFTEEYQL FEELGKGAFS VVRRCVKVLA GQEYAAKIIN TKKLSARDHQ KLEREARICR LLKHPNIVRL HDSISEEGHH YLIFDLVTG GELFEDIVAR EYYSEADASH CIQQILEAVL HCHQMGVVHR DLKPENLLLA SKLKGAAVKL ADFGLAIEVE G EQQAWFGF AGTPGYLSPE VLRKDPYGKP VDLWACGVIL YILLVGYPPF WDEDQHRLYQ QIKAGAYDFP SPEWDTVTPE AK DLINKML TINPSKRITA AEALKHPWIS HRSTVASCMH RQETVDCLKK FNARRKLKGA ILTTMLATRN FSGGKSGGNK KSD GVKESS ESTNTTIEDE DTKVRKQEII KVTEQLIEAI SNGDFESYTK MCDPGMTAFE PEALGNLVEG LDFHRFYFEN LWSR NSKPV HTTILNPHIH LMGDESACIA YIRITQYLDA GGIPRTAQSE ETRVWHRRDG KWQIVHFHRS GAPSVLPH UniProtKB:  Calcium/calmodulin-dependent protein kinase type II subunit alpha Calcium/calmodulin-dependent protein kinase type II subunit alpha |
-分子 #2: Calcium/calmodulin-dependent protein kinase type II subunit alpha
| 分子 | 名称: Calcium/calmodulin-dependent protein kinase type II subunit alpha タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号:  Ca2+/calmodulin-dependent protein kinase Ca2+/calmodulin-dependent protein kinase |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 53.634984 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: STRFTEEYQL FEELGKGAFS VVRRCVKVLA GQEYAAMIIN TKKLSARDHQ KLEREARICR LLKHPNIVRL HDSISEEGHH YLIFDLVTG GELFEDIVAR EYYSEADASH CIQQILEAVL HCHQMGVVHR NLKPENLLLA SKLKGAAVKL ADFGLAIEVE G EQQAWFGF ...文字列: STRFTEEYQL FEELGKGAFS VVRRCVKVLA GQEYAAMIIN TKKLSARDHQ KLEREARICR LLKHPNIVRL HDSISEEGHH YLIFDLVTG GELFEDIVAR EYYSEADASH CIQQILEAVL HCHQMGVVHR NLKPENLLLA SKLKGAAVKL ADFGLAIEVE G EQQAWFGF AGTPGYLSPE VLRKDPYGKP VDLWACGVIL YILLVGYPPF WDEDQHRLYQ QIKAGAYDFP SPEWDTVTPE AK DLINKML TINPSKRITA AEALKHPWIS HRSTVASCMH RQETVDCLKK FNARRKLKGA ILTTMLATRN FSGGKSGGNK KSD GVKESS ESTNTTIEDE DTKVRKQEII KVTEQLIEAI SNGDFESYTK MCDPGMTAFE PEALGNLVEG LDFHRFYFEN LWSR NSKPV HTTILNPHIH LMGDESACIA YIRITQYLDA GGIPRTAQSE ETRVWHRRDG KWQIVHFHRS GAPSVLPH UniProtKB:  Calcium/calmodulin-dependent protein kinase type II subunit alpha Calcium/calmodulin-dependent protein kinase type II subunit alpha |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: C-flat-1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 詳細: 30 s using PELCO easiGlow |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 96 % / 装置: GATAN CRYOPLUNGE 3 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: OTHER |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 52.0 e/Å2 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


























 Z
Z Y
Y X
X